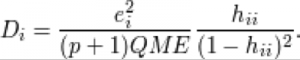Ferramentas do usuário
Barra lateral
Tabela de conteúdos
Testes Clássicos
Anova
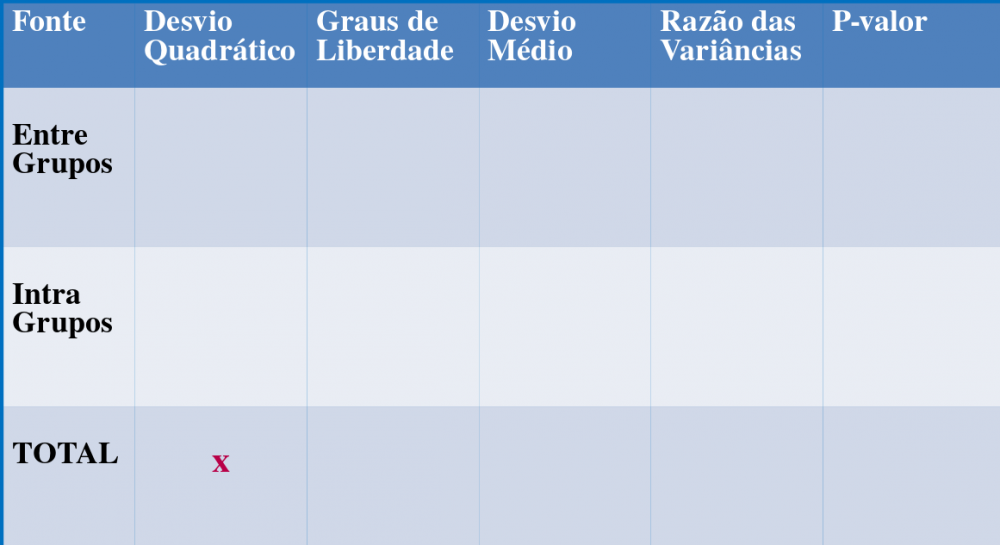
Tabela de Anova
Baixe o arquivo colheita.csv e preencha a tabela de anova com esses dados. Testando a hipótese de que existem diferenças na produção agrícola em diferentes tipos de solo. Os cálculos devem ser feitos passo-a -passo, sem uso de uma função específica.
Desvios quadráticos total
$$SS_{total} = \sum_{i=1}^k\sum_{j=1}^n (y_{ij} - \bar{\bar{y}})^2 $$
Desvios quadráticos internos ao grupo
$$SS_{in} = \sum_{i=1}^k\sum_{j=1}^n (y_{i,j} - \bar{y}_{i})^2 $$
Desvio quadrático entre os grupos
$$SS_{en} = \sum_{i=1}^k\sum_{j=1}^n (\bar{y}_{i} - \bar{\bar{y}})^2 $$
- O resto é com vc, COMPLETE A TABELA!
- Faça o teste usando reamostragem no RSampling
- Faça gráficos para apresentar os dados
- Inclua o resultado do teste de ANOVA no gráfico
Regressão Linear Simples
Análise de Resíduos de Regressão Linear
Quando realizamos uma análise mais aprofundada sobre a relação entre duas variáveis numéricas contínuas podemos ajustar uma reta que represente o melhor ajuste entre os dados e que possa nos ajudar a prever valores da variável resposta (eixo Y) a partir de valores da variável preditora (eixo X). Se o ajuste é uma reta, esse tipo de análise é chamado de Análise de Regressão Linear. A explicação sobre como funciona essa análise foi apresentada na aula sobre Análise de Regressão Linear. Alguns aspectos importantes para entendermos esse tutorial:
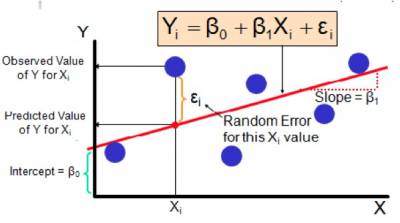
- A reta ajustada (também denominada “linha de regressão”) passa obrigatoriamente pelo ponto que representa a média da variável Y e a média da variável X.
- A linha de regressão é aquela que minimiza os resíduos (na verdade, a soma dos quadrados dos resíduos)
- Os pontos das observações estarão distribuídos em torno dessa reta.
- A distância vertical (projetada no eixo Y) de cada ponto até a reta é chamada de resíduo ou erro dos pontos.
Nesse tutorial nosso interesse é avaliar como os resíduos/erros estão distribuídos, pois os modelos de regressão linear possuem importantes premissas relacionadas a eles.
As premissas de um modelo de regressão linear são relativos aos termos de resíduos/erros do modelo. Se estamos falando de um modelo de regressão no qual a variável preditora é fixa (i.e. sem erros aleatórios), somente a variável resposta apresentará erro aleatório, então, as premissas também se aplicam à variável Y (resposta).
Premissas de uma Análise de Regressão Linear
- LINEARIDADE - Uma reta representa o melhor ajuste aos dados
- DISTRIBUIÇÃO NORMAL DOS ERROS/RESÍDUOS - Para cada valor de X, os erros seguem uma distribuição normal. Se fossem feitas muitas réplicas para cada um dos valores de X, a distribuição dos vários valores obtidos para Y (e consequentemente dos erros) nas muitas réplicas seguiria uma distribuição normal. Porém, em geral, não são feitas réplicas e é necessário assumir que esses valores seguem essa distribuição.
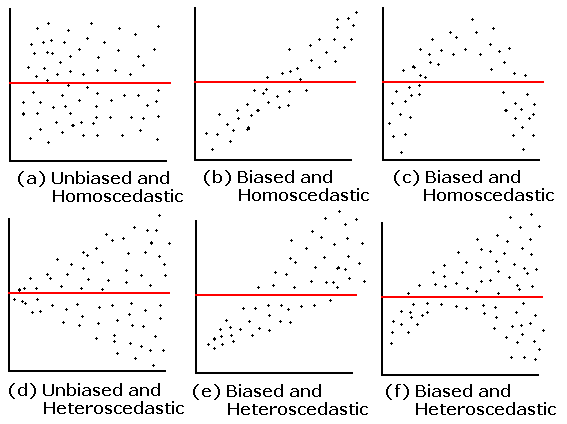
- VARIÂNCIA DOS ERROS/RESÍDUOS CONSTANTE - Para qualquer valor de X, a variância dos erros é a mesma. Se fossem feitas muitas réplicas para cada um dos valores de X, a distribuição dos vários valores obtidos para Y (e consequentemente dos erros) nas muitas réplicas apresentaria uma mesma variância para qualquer valor de X. Porém, em geral, não são feitas réplicas e é necessário assumir que essas variâncias são iguais.
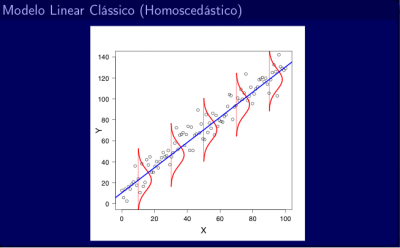
Quando essa premissa é cumprida, temos o que chamamos de homoscedasticidade:
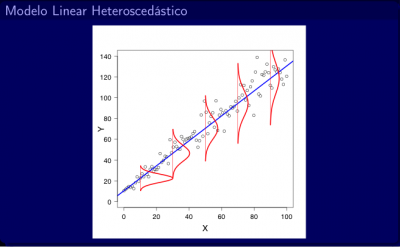
Quando ela não é cumprida, observamos uma heteroscedasticidade:
O que são os erros/resíduos e como calcular?
Os erros/resíduos indicam o quão longe os valores de Y observados estão dos valores de Y estimados pela linha de regressão ajustada.
Os erros/resíduos de cada observação são calculados projetando-se no eixo Y o valor de Y observado e o valor de Y estimado (ou predito) pela reta e calculando-se a diferença entre esses dois valores.
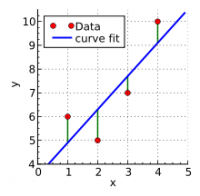
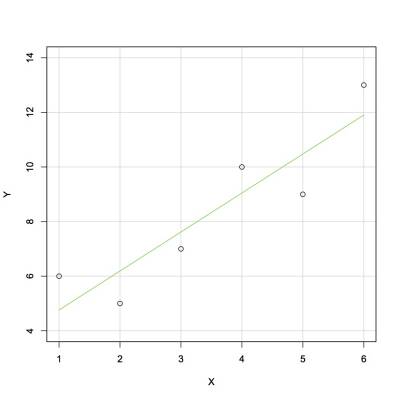
Agora vamos estimar os valores dos resíduos para esse exemplo hipotético abaixo:
Faça uma tabela como essa e anote os valores aproximados que você consegue obter por esse gráfico:
| X | Yobservado | Yestimado | Resíduo |
|---|---|---|---|
| 1 | |||
| 2 | |||
| 3 | |||
| 4 | |||
| 5 | |||
| 6 |
Agora vamos checar no R com esses mesmos dados: 1) Crie um diretório (i.e. uma pasta) para você
2) Abra o R no seu computador e mude o diretório de trabalho para o diretório que você criou, usando o menu Arquivo > mudar dir….
3) Crie as variáveis x e y:
x<- c(1,2,3,4,5,6) y<- c(6,5,7,10,9,13)
4) Ajuste um modelo de regressão linear simples usando a função lm() e inspecione o resumo do modelo usando a função summary(), que fornece informações importantes sobre o modelo, incluindo os valores brutos dos erros/resíduos (residuals):
lm.xy<-lm(y~x) summary(lm.xy)
Checando as premissas
Ok, agora que você entendeu como são calculados os erros/resíduos, vamos trabalhar com conjuntos de dados maiores para podermos entender como checar as premissas da análise de regressão linear de uma maneira um pouco mais realista:
Baixe os arquivos de dados para o seu diretório:
Descrição dos conjuntos de dados:
Um grupo de pesquisadores vem trabalhando há muito tempo com peixes da família Rivulidae que ocorrem em lagos temporários. Esses peixes crescem e se reproduzem nesses lagos temporários durante o período de chuvas e seus ovos ficam dormentes durante o período de seca.
Do total de lagos temporários existentes, foram sorteados 20 lagos e na época chuvosa os seguintes dados foram coletados:
- - Biomassa de algas
- - Biomassa de insetos aquáticos
- - Volume do lago
- - Biomassa de peixes herbívoros
- - Biomassa de peixes insetívoros
- - Número de indivíduos adultos da espécie mais abundante (Austrolebias charrua)
- O primeiro conjunto de dados (algas_peixes.csv) foi obtido com o objetivo de analisar se a biomassa de algas existente nos lagos influencia a biomassa de peixes herbívoros e se essa relação é linear.
- O segundo conjunto de dados (algas_peixes2.csv) foi obtido com o mesmo objetivo anterior, mas em outros 20 lagos diferentes
- O terceiro conjunto de dados (insetos_peixes.csv) foi obtido com o objetivo de analisar se a biomassa de insetos existente nos lagos influencia a biomassa de peixes insetívoros e se essa relação é linear.
- O quarto conjunto de dados (vol_inds.csv) foi obtido com o objetivo de analisar se o volume de água de cada lago afeta o número de indivíduos da espécie Austrolebias charrua existente no lago e se essa relação é linear.
Carregue o pacote car:
library(car)
O primeiro passo é ajustar um modelo de regressão linear aos dados obtidos.
Inicialmente vamos trabalhar com o conjunto de dados algas_peixes.csv
Importe o arquivo para o R e conheça os dados:
algas.peixes <- read.csv("algas_peixes.csv", sep=";")
head(algas.peixes)
summary(algas.peixes)
Avalie visualmente a relação entre as variáveis com o gráfico scatterplot:
scatterplot(BIOMASSA_PEIXES_HERB~BIOMASSA_ALGAS, data=algas.peixes)
Ajuste um modelo de regressão linear para as variáveis, usando a função lm():
lm.algas.peixes<-lm(BIOMASSA_PEIXES_HERB~BIOMASSA_ALGAS, data=algas.peixes) summary (lm.algas.peixes)
Use a função “names()” para saber quais são as informações que estão disponíveis sobre esse modelo:
names(lm.algas.peixes)
Se você quiser olhar detalhadamente alguma dessas informações, basta escrever o nome_do_modelo$nome_da_informação. Então, vamos olhar especificamente os erros/resíduos:
lm.algas.peixes$residuals
O mesmo pode ser feito para conhecer os valores ajustados (fitted.values), os coeficientes a e b (coef), etc.
Como saber se os erros/resíduos seguem uma distribuição normal?
Lembre dos métodos usados no tutorial de ANÁLISES EXPLORATÓRIAS DE DADOS. Escolha um dos métodos disponíveis para avaliar a normalidade dos dados e aplique a mesma lógica para a distribuição dos erros/resíduos.
Histograma
hist(lm.algas.peixes$residuals)
Boxplot
boxplot(lm.algas.peixes$residuals)
Gráfico Quantil-Quantil
qqnorm(lm.algas.peixes$residuals) qqline(lm.algas.peixes$residuals)
Como saber se a variância dos erros/resíduos é constante?
Para qualquer valor de X (ou de Yobservado, ou de Yestimado) os valores máximos e mínimos dos resíduos devem ser similares. Então, podemos fazer um gráfico em que relacionamos os valores de Yestimado (ou seja, os valores de Y que são indicados pela reta de regressão) e os valores dos Resíduos para cada Yestimado.
res.a.p<-lm.algas.peixes$residuals
yest.a.p<-lm.algas.peixes$fitted.values
plot(res.a.p~yest.a.p, xlab="Y estimado", ylab="Resíduos")
Como você interpreta esse gráfico? Você nota algum padrão na distribuição dos erros/resíduos?
O mesmo gráfico (Resíduos X Yestimado) que é utilizado para avaliar se a variância é constante (homoscedasticidade), também pode ser utilizado para checar se existe alguma assimetria, algum viés (positivo ou negativo) ou alguma tendência de que a relação seja melhor definida por uma curva do que por uma reta.
A figura abaixo mostra vários exemplos desse gráfico entre Resíduos X Yestimado relações com ou sem homoscedasticidade e com ou sem vieses (biased ou unbiased):
Como saber se uma reta representa o melhor ajuste?
O primeiro gráfico a ser feito é um gráfico de dispersão (XY) simples. Uma curva suavizada pode ser plotada para ajudar a analisar a tendência geral.
scatterplot(y~x)
Adicionalmente, o gráfico de Resíduos X Yestimado (acima) também indica se existe alguma tendência de melhor ajuste a uma curva do que a uma reta.
Como saber se alguma observação está influenciando demais os parâmetros da regressão?
Além de testar as premissas, também é importante fazer um diagnóstico para verificar se existem outliers e se eles afetam muito o resultado da análise de regressão.
Para medir a influência de uma observação usamos uma medida denominada “Distância de Cook” que é calculada para cada observação e leva em consideração o erro/resíduo (e) e a leverage (hii) da observação, que pode ser traduzida como “alavancagem”. A leverage indica o quanto um dado valor de X influencia o valor de Yestimado.
Valores altos de Distância de Cook significam que se esse ponto for retirado das análises, a inclinação da reta de regressão pode mudar muito. Veja o exemplo abaixo, do livro de Quinn & Keough (2008), mostrando o efeito de três diferentes pontos sobre a inclinação da reta.
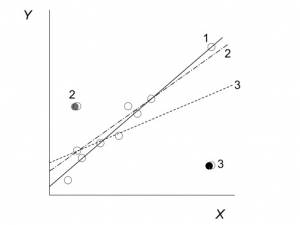
Se o valor dos Resíduos for plotado em relação ao valor de leverage, os pontos que possuírem as maiores leverage e também erros/resíduos grandes (positivos ou negativos) serão os pontos com maiores Distâncias de Cook e consequentemente, com maiores influências sobre os parâmetros da reta.
Devido ao tempo escasso, não vamos construir esse gráfico passo-a-passo. Vamos usar uma função mágica do R que vai mostrar 4 gráficos de diagnóstico de uma só vez e incluirá esse gráfico para que você possa analisar.
O primeiro passo é ajustar um modelo de regressão linear aos dados obtidos. Para o primeiro conjunto de dados (algas_peixes.csv), nós já fizemos isso, então, vamos apenas inspecionar o resumo do modelo:
summary (lm.algas.peixes)
Agora, vamos definir que sejam construídos os 4 gráficos de diagnóstico para esse modelo e que eles sejam colocados em uma mesma página:
par(mfrow=c(2,2)) plot(lm.algas.peixes) par(mfrow=c(1,1))
Obs.: Note que o gráfico inferior à direita é o gráfico que mostra a distância de Cook.
Salve essa página como.pdf e coloque o mesmo nome do arquivo de dados
Repita o mesmo procedimento para os outros conjuntos de dados e avalie quais premissas estão sendo atendidas ou não para cada um.
## copie uma linha por vez:
algas.peixes2 <- read.csv("algas_peixes2.csv", sep=";")
head(algas.peixes2)
summary(algas.peixes2)
scatterplot(BIOMASSA_PEIXES_HERB2~BIOMASSA_ALGAS2, data=algas.peixes2)
lm.algas.peixes2<-lm(BIOMASSA_PEIXES_HERB2~BIOMASSA_ALGAS2, data=algas.peixes2)
summary (lm.algas.peixes2)
## copie as três linhas juntas:
par(mfrow=c(2,2))
plot (lm.algas.peixes2)
par(mfrow=c(1,1))
## copie uma linha por vez:
insetos.peixes <- read.csv("insetos_peixes.csv", sep=";")
head(insetos.peixes)
summary(insetos.peixes)
scatterplot(BIOMASSA_PEIXES_INS~BIOMASSA_INSETOS, data=insetos.peixes)
lm.insetos.peixes<-lm(BIOMASSA_PEIXES_INS~BIOMASSA_INSETOS, data=insetos.peixes)
summary(lm.insetos.peixes)
## copie as três linhas juntas:
par(mfrow=c(2,2))
plot (lm.insetos.peixes)
par(mfrow=c(1,1))
## copie uma linha por vez:
vol.inds <- read.csv("vol_inds.csv", sep=";")
head(vol.inds)
summary(vol.inds)
scatterplot(INDIVIDUOS_AUSTROL~VOLUME_LAGO, data=vol.inds)
lm.vol.inds<-lm(INDIVIDUOS_AUSTROL~VOLUME_LAGO, data=vol.inds)
summary(lm.vol.inds)
## copie as três linhas juntas:
par(mfrow=c(2,2))
plot (lm.vol.inds)
par(mfrow=c(1,1))