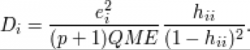Ferramentas do usuário
Barra lateral
Tabela de conteúdos
Testes Clássicos
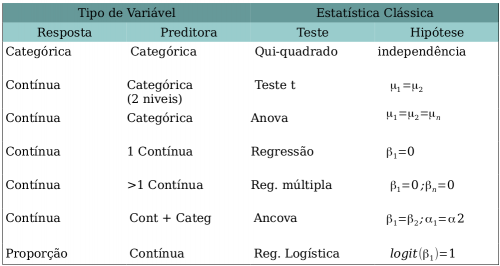
Os testes clássicos estatísticos estão inseridos no escopo da estatística frequentista ou inferência frequentista. Nessa abordagem a probabilidade é considerada uma frequência e a inferencia está baseada na frequência com que eventos ocorrem nos dados coletados. A maior parte dos testes frequentistas clássicos foi desenvolvida independentemente para a solução de problemas distintos. Por isso, não há uma unificação analítica completa, que só aconteceu posteriormente com a integração oferecida pelos modelos lineares, como veremos nas próximas aulas. Nos testes clássicos a aplicação é definida basicamente pela natureza das variáveis resposta (dependente) e preditora (independente) e pela hipótese estatística subjacente.
Regressão Linear Simples
Conceitos importantes
Quando realizamos uma análise mais aprofundada sobre a relação entre duas variáveis numéricas contínuas podemos ajustar uma reta que represente o melhor ajuste entre os dados e que possa nos ajudar a prever valores da variável resposta (eixo Y) a partir de valores da variável preditora (eixo X). Se o ajuste é uma reta, esse tipo de análise é chamado de Análise de Regressão Linear. A explicação detalhada sobre como funciona essa análise foi apresentada na aula sobre Análise de Regressão Linear. Alguns aspectos importantes que precisam ser lembrados para entendermos esse tutorial:
- A reta ajustada (também denominada “linha de regressão”) passa obrigatoriamente pelo ponto que representa a média da variável Y e a média da variável X.
- Os pontos das observações estarão distribuídos em torno dessa reta.
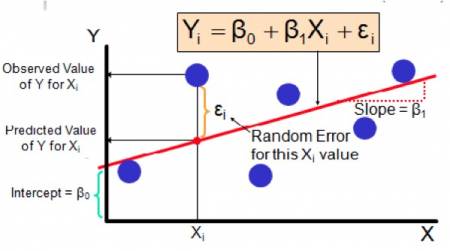
- A relação (reta) entre a variável preditora (x) e a variável resposta (y) é descrita por uma equação simples (y=a+bx) com apenas dois parâmetros: a (intercepto) e b (inclinação da reta).
- O intercepto (a) representa o valor estimado da variável resposta (y) quando a variável preditora (x) é igual à zero.
- A inclinação (b) representa o efeito da variável preditora (x) sobre a variável resposta (y), ou seja, o quanto a variável y aumenta (ou diminui) à cada unidade da variável preditora (x).
- Quando usamos regressão linear para testar hipóteses científicas em ecologia, majoritariamente estamos interessados em saber o efeito da variável preditora (x) sobre a variável resposta (y), ou seja, se a inclinação da reta (b) é significativamente diferente de zero. Em geral, não temos hipóteses científicas acerca do intercepto do modelo.
- A distância vertical (projetada no eixo Y) de cada ponto até a reta é chamada de resíduo, desvio ou erro daquele ponto.
- A linha de regressão é aquela que minimiza os resíduos (na verdade, a soma dos quadrados dos resíduos)
O objetivo desse tutorial é fazer e interpretar os resultados de uma análise de regressão linear, assim como avaliar os pressupostos do modelo.
O que são os erros/resíduos e como calcular?
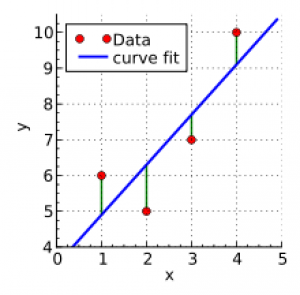
Os erros/resíduos indicam o quão longe os valores de Y observados estão dos valores de Y estimados pela linha de regressão ajustada. Eles estão representados em verde na figura abaixo:
Os erros/resíduos de cada observação são calculados projetando-se no eixo Y o valor de Y observado e o valor de Y estimado (ou predito) pela reta e calculando-se a diferença entre esses dois valores.
Premissas de uma Análise de Regressão Linear
- LINEARIDADE - Uma reta representa o melhor ajuste aos dados
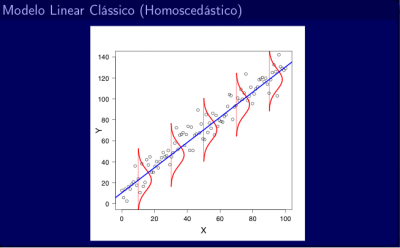
- DISTRIBUIÇÃO NORMAL DOS ERROS/RESÍDUOS - Para cada valor de X, os erros devem seguir uma distribuição normal. Se fossem feitas muitas réplicas para cada um dos valores de X, a distribuição dos erros referentes aos vários valores obtidos para Y nas muitas réplicas seguiria uma distribuição normal (Veja as curvas em vermelho na figura de homoscedasticidade abaixo). Porém, em geral, não são feitas réplicas e é necessário assumir que os resíduos seguem essa distribuição.
- VARIÂNCIA DOS ERROS/RESÍDUOS CONSTANTE - Para qualquer valor de X, a variância dos erros é a mesma. Se fossem feitas muitas réplicas para cada um dos valores de X, a distribuição dos erros referentes aos vários valores obtidos para Y nas muitas réplicas apresentaria uma mesma variância para qualquer valor de X. Porém, em geral, não são feitas réplicas e é necessário assumir que essas variâncias são iguais.
Quando essa premissa é cumprida, temos o que chamamos de homoscedasticidade:
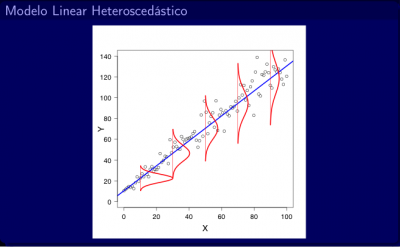
Quando ela não é cumprida, observamos uma heteroscedasticidade. Note na figura abaixo que para valores pequenos de X a variância é menor (distribuição estreita) e que para valores maiores de X temos uma variância grande (distribuição larga):
Agora vamos checar no R com esses mesmos dados: 1) Crie um diretório (i.e. uma pasta) para você
2) Abra o R no seu computador e mude o diretório de trabalho para o diretório que você criou, usando o menu Arquivo > mudar dir….
3) Crie as variáveis x e y:
x<- c(1,2,3,4,5,6) y<- c(6,5,7,10,9,13)
4) Ajuste um modelo de regressão linear simples usando a função lm() e inspecione o resumo do modelo usando a função summary(), que fornece informações importantes sobre o modelo, incluindo os valores brutos dos erros/resíduos (residuals):
lm.xy<-lm(y~x) summary(lm.xy)
Checando as premissas
Ok, agora que você entendeu como funciona a regressão linear, vamos para um exemplo prático.
Baixe o arquivo de dados para o seu diretório:
- produtividade_chuva.txt|chuva.txt}}
Descrição dos conjuntos de dados:
Atenção: esses conjuntos de dados não são reais, são simulações produzidas com o objetivo pedagógico.
Pesquisadores interessados em entender o efeito da precipitação sobre a produtividade primária liquída em ecossistemas terrestres, selecionaram 30 áreas naturais distribuídas por todo o globo em diferentes ecossistemas. Dada a importância da água para a fotossíntese, a hipótese dos pesquisadores era que quanto maior a precipitação, maior seria a produtividade primária líquida do ecossistemas. Em cada área, os pequisadores coletaram duas informações: a precipitação anual média (mm) e a produtividade primária líquida (Mg/ha/ano).
Hipóteses estatísticas
Considerando que os pesquisadores estão interessados no efeito da precipitação sobre a produtividade, podemos assumir que a precipitação é a variável preditora (x) e a produtividade é a variável resposta (y). Como ambas as variáveis são contínuas, podemos aplicar uma regressão linear simples para testar a hipótese científica. Neste caso, o efeito de precipitação sobre a produtividade será descrito pela inclinação da reta (b). Sendo assim, as hipóteses estatísticas serão:
- H0: B=0
- H1: B≠0
Carregue o pacote car:
library(car)
Importe o arquivo para o R e conheça os dados:
algas.peixes <- read.csv("algas_peixes.csv", sep=";")
head(algas.peixes)
summary(algas.peixes)
Avalie visualmente a relação entre as variáveis com o gráfico scatterplot:
scatterplot(BIOMASSA_PEIXES_HERB~BIOMASSA_ALGAS, data=algas.peixes)
Ajuste um modelo de regressão linear para as variáveis, usando a função lm():
lm.algas.peixes<-lm(BIOMASSA_PEIXES_HERB~BIOMASSA_ALGAS, data=algas.peixes) summary (lm.algas.peixes)
Use a função “names()” para saber quais são as informações que estão disponíveis sobre esse modelo:
names(lm.algas.peixes)
Se você quiser olhar detalhadamente alguma dessas informações, basta escrever o nome_do_modelo$nome_da_informação. Então, vamos olhar especificamente os erros/resíduos:
lm.algas.peixes$residuals
O mesmo pode ser feito para conhecer os valores ajustados (fitted.values), os coeficientes a e b (coef), etc.
Como saber se os erros/resíduos seguem uma distribuição normal?
Base
Os testes clássicos estatísticos estão inseridos no escopo da estatística frequentista ou inferência frequentista. Nessa abordagem a probabilidade é considerada uma frequência e a inferencia está baseada na frequência com que eventos ocorrem nos dados coletados. A maior parte dos testes frequentistas clássicos foi desenvolvida independentemente para a solução de problemas distintos. Por isso, não há uma unificação analítica completa, que só aconteceu posteriormente com a integração oferecida pelos modelos lineares, como veremos nas próximas aulas. Nos testes clássicos a aplicação é definida basicamente pela natureza das variáveis resposta (dependente) e preditora (independente) e pela hipótese estatística subjacente.
Anova
Aula Gravada - Anova: Partição da Variaçao
Esssa video aula foi gravada durante a pandemia e permanece aqui como material de referência e consulta
Na aula sobre teste de hipótese utilizamos técnicas de Monte Carlo para testar a hipótese de que duas médias são distintas, ou que uma é maior/menor que outra, tanto no exemplo do Tutorial Árvores do Mangue, quanto no exercício Altura dos alunos. Em ambos os casos estávamos comparando médias de dois grupos distintos, por exemplo, dois tipos de solos no mangue ou gênero dos alunos. O nosso procedimento foi análogo ao teste frequentista t de Student, mas a forma de obter o p-valor foi diferente. Nos procedimentos anteriores, simulamos o cenário nulo e comparamos o valor observado (diferença das médias) com a distribuição de probabilidades obtidas por meio dessa simulação. Na abordagem clássica do teste frequentista t de Student, a estatística de interesse t da amostra é comparada com a distribuição probabilística t , desenvolvida pelo matemático britânico William Gosset.
Caso não esteja confortável com o procedimento de simulação do cenário nulo e consequente obtenção do p-valor , refaça o tutorial teste de hipótese. No procedimento apresentado está a lógica básica por trás da maioria dos testes de hipótese clássicos.
A Análise de Variância ( ANOVA ), desenvolvida pelo também britânico Ronald Fisher há mais de 100 anos (1918), é uma generalização do teste t de Student. Apesar da idade avançada, é um teste muito popular, talvez o mais utilizado em ciências naturais nas últimas décadas. A hipótese subjacente da ANOVA é de diferença entre as médias de 2 ou mais grupos. O procedimento para o cálculo da estatística da ANOVA, chamada de F, está associado à partição da variância dos dados, por isso o nome. Uma maneira clássica de apresentar o resultado do teste de ANOVA é a a chamada tabela de ANOVA. Tanto a partição da variação quanto a tabela de ANOVA serão utilizados para avaliarmos outros modelos durante o curso, por isso é importante entender bem o que é a partição da variação e o que a tabela de ANOVA nos apresenta.
Partição da Variância
O teste de ANOVA está baseado na premissa de que os efeitos entre os grupos são aditivos e com isso é possível particionar a variação dos dados na porção que é associada aos grupos e a que representa a variação não explicada (resíduos ou erros). A soma destas variações resultam na variação total associada aos dados.
Para exemplificar a partição da variância associada à ANOVA, vamos usar o exemplo de dados de colheita de um cultivar em diferentes tipos de solos, apresentado no livro de Robert Crawley, The R Book, como segue abaixo:
Tradução livre da descrição do livro “The R Book” (Crawley, 2007)
“… a melhor forma de entender o que está acontecendo é trabalharmos um exemplo. Temos um experimento em que a produção agrícola por unidade de área é medida em 10 campos de cultivo selecionados aleatoriamente de cada um de três tipos diferentes de solo. Todos os campos foram semeados com a mesma variedade de semente e manejados com as mesmas técnicas (fertilizantes, controle de pragas). O objetivo é verificar se o tipo de solo afeta significativamente o rendimento de culturas, e caso afete, quanto.”1)
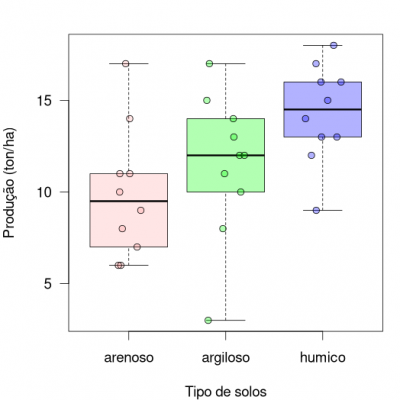
A representação gráfica desses dados pode ser feita em um boxplot.
É possível notar que há uma grande variação na produtividade entre os solos e também muita variação dentro de um mesmo tipo de solo. Para ter alguma confiança para afirmar que o solo influencia a produtividade, podemos nos basear na variação dos dados e na partição em seus componentes, ou seja, dentro de cada grupo (ou intra grupo) e entre os grupos do tratamento (tipos de solos). Primeiro vamos definir o que é a variação total dos dados.
Variação total
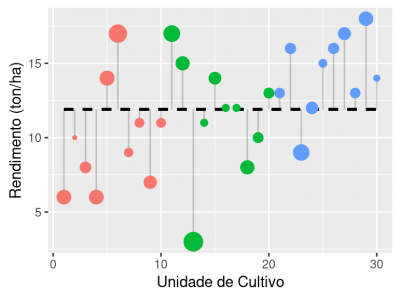 A variação total dos dados é calculada a partir dos desvios das observações (n=30) em relação à grande média 2)). No gráfico acima esta variação é representada pelos segmentos verticais em cinza. A grande média é definida como a média de produtividade de todos os campos de cultivo, independente do tipo de solo, e é representada pela linha preta horizontal tracejada.
A variação total dos dados é calculada a partir dos desvios das observações (n=30) em relação à grande média 2)). No gráfico acima esta variação é representada pelos segmentos verticais em cinza. A grande média é definida como a média de produtividade de todos os campos de cultivo, independente do tipo de solo, e é representada pela linha preta horizontal tracejada.
Medimos essa variação total pela soma quadrática definida como os valores dos desvios dos dados em relação à grande média (segmentos verticais no gráfico) elevados ao quadrado e posteriormente somados.
$$ SQ_{"total"} = \sum_{i=1}^k\sum_{j=1}^n (y_{ij} - \bar{\bar{y}})^2 $$
Variação intra grupo
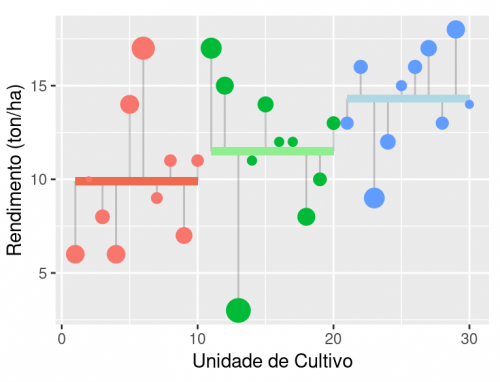 A variação intra grupo é a variação que não está relacionada ao efeito do tratamento (no caso, os tipos de solo). Essa variação é baseada nos desvios dos valores observados em relação à média do nível de tratamento (tipo de solo ou grupo) representada pelos segmentos horizontais coloridos. Os respectivos desvios estão representados na figura acima pelas barras cinza verticais.
A variação intra grupo é a variação que não está relacionada ao efeito do tratamento (no caso, os tipos de solo). Essa variação é baseada nos desvios dos valores observados em relação à média do nível de tratamento (tipo de solo ou grupo) representada pelos segmentos horizontais coloridos. Os respectivos desvios estão representados na figura acima pelas barras cinza verticais.
Entendemos desvios como qualquer variação em relação a alguma medida de tendencia central, no caso estamos tratando dessa variação em relação a diferentes médias (grande média e média dos grupos). Mais à frente iremos chamar esses desvios das observações em relação às médias do seu grupo de resíduos e muitos estatísticos também os chamam essa variação de erro. Não se assustem, eles significam a mesma coisa e causam confusão, mesmo. O importante é entender que estamos nos referindo à variação que não é explicada pelos tratamentos.
Para quantificar essa variação utilizamos a soma quadrática intra grupo, obtida a partir desses valores de desvios 3). Basta pegar a diferença entre cada valor observado em relação à média do seu grupo, elevar ao quadrado e posteriormente somar esses valores, como descrito na formula a seguir:
$$ SQ_{"intra"} = \sum_{i=1}^k\sum_{j=1}^n (y_{i,j} - \bar{y}_{i})^2 $$
Variação entre grupos
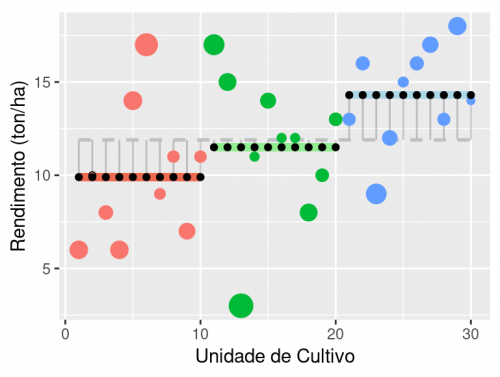 Por fim, temos a variação entre os grupos. Essa variação está diretamente relacionada ao efeito dos níveis do nosso tratamento, que no caso são os tipos de solo. Ou seja, quanto maior o efeito do tipo de solo na produtividade, maior será essa variação. Ela é definida pelos desvios das médias dos grupos em relação à grande média (segmentos verticais cinzas). Essa variação pode ser representada substituindo cada valor observado (círculos coloridos) pela média do seu grupo (círculos pretos). Os desvios desses valores médios dos grupos em relação à grande média, elevado ao quadrado e somados, representam a soma quadrática entre grupos.
Por fim, temos a variação entre os grupos. Essa variação está diretamente relacionada ao efeito dos níveis do nosso tratamento, que no caso são os tipos de solo. Ou seja, quanto maior o efeito do tipo de solo na produtividade, maior será essa variação. Ela é definida pelos desvios das médias dos grupos em relação à grande média (segmentos verticais cinzas). Essa variação pode ser representada substituindo cada valor observado (círculos coloridos) pela média do seu grupo (círculos pretos). Os desvios desses valores médios dos grupos em relação à grande média, elevado ao quadrado e somados, representam a soma quadrática entre grupos.
$$ SQ_{"entre"} = \sum_{i=1}^k\sum_{j=1}^n (\bar{y}_{i} - \bar{\bar{y}})^2 $$
Variação aditiva
Acabamos de particionar a variação dos dados de um teste de ANOVA em seus componentes básicos: a variação entre e intra grupos. Uma característica importante dessa partição é que suas partes são aditivas, ou seja, a variação total é a soma da intra e entre grupos.
$$ SQ_{"total"} = SQ_{"entre"} + SQ_{"intra"} $$
Estatística F
A grande sacada de Sir Fisher foi entender que essa partição da variância aditiva pode ser utilizada para compor uma estatística que representa o quanto a variação do efeito do tratamento é maior que a variação não explicada pelo tratamento. A estatística F é definida pela razão do valor médio da variação entre grupos e o valor médio da variação intra grupos.
Os valores médios de variação (variância) são calculados dividindo as somas quadráticas pelos graus de liberdade. No caso da variação entre grupos do nosso exemplo o total de graus de liberdades é igual ao número de grupos no tratamento menos 1 (em função do parâmetro média geral usado para o seu cálculo). Na variação intra grupos o total de graus de liberdade é igual ao número de observações (30 valores usados para o seu cálculo) menos 3 (número de parâmetros utilizados para o seu cálculo, as médias dos grupos), no caso 27.
$$MQ_{"entre"} = \frac{SQ_{"entre"}}{gl_1}$$
$$MQ_{"intra"} = \frac{SQ_{"intra"}}{gl_2}$$
$$F_{(gl_1,gl_2)} = \frac{MQ_{1}}{MQ_{2}}$$
sendo:
- F: estatística F
- gl: graus de liberdade
- ${_1}$: entre grupos
- ${_2}$: intra grupos
A probabilidade de ocorrência de valores da estatística F sob um cenário nulo segue uma distribuição desenvolvida por Sir Ronald Fisher e por George Snedecor. Essa distribuição possui dois parâmetros, os graus de liberdade entre e intra grupos. Assim, para calcular o p-valor para um dado valor de F observado no nosso estudo, usamos os graus de liberdade entre grupos e os graus de liberdade intra grupos para consultarmos uma tabela de F ou utilizarmos algum programa que tenha essa distribuição definida.
Coeficiente de determinação
Outra estatística muito utilizada, baseada na partição de variação, é o coeficiente de determinação. O coeficiente de determinação define o quanto da variabilidade dos dados é explicado pelo fator de interesse, no nosso exemplo, os tipos de solos. O coeficiente de determinação (R²) é calculado pela razão entre a variação explicada e a variação total dos dados.
$$ R^2 = \frac{SQ_{"entre"}}{SQ_{"entre"} + SQ_{"intra"}} $$
Tabela de ANOVA
Para fixar esses conceitos vamos construir uma tabela de ANOVA em uma planilha de Excel ou LibreOffice.
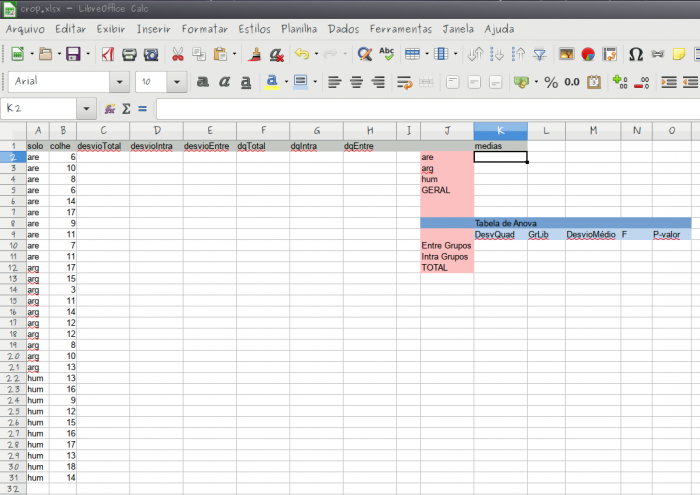
- baixe o arquivo crop.xlsx;
- abra em uma planilha eletrônica;
- 1) a partir dos dados de produtividade (colheita) obtidos, calcule a média de cada grupo e a média geral e guarde nas células correspondentes à direita, na coluna K;
- 2) na coluna “desvioTotal” calcule o quanto cada observação desvia da média geral;
- 3) na coluna “desvioIntra” calcule o quanto cada observação desvia da média do seu grupo;
- 4) na coluna “desvioEntre” calcule, para cada observação, o quanto a média do seu grupo desvia da média geral;
- 5) nas colunas de desvio quadrático (dq*) correspondentes a cada coluna anterior, eleve ao quadrado cada um dos desvios calculados anteriormente.
- O que representam as somas das colunas (dq*)?
- 6) usando as orientações acima e as informações fornecidas na aula complete a tabela de ANOVA;
- 7) usando a dica abaixo, calcule o p-valor e insira na tabela de ANOVA;
Como calcular o p-valor a partir do F
- A função
DIST.Fno Excel ou LibreOffice calcula o p-valor a partir da estatística F e graus de liberdade; - usualmente a função recebe o valor de F, seguido dos graus de liberdade entre e intra grupos;
- normalmente o resultado da função
DIST.Fé a probabilidade cumulativa, mas fique atento, pode ser a densidade probabilística, dependendo do padrão do Excel. Consulte a documentação do ''DIST.F'' do Excel caso tenha dúvida; - no caso do valor retornado seja a probabilidade cumulativa, o p-valor é igual a 1 menos essa probabilidade 4).
- 8) a partir das dicas abaixo, repita o teste no Rcmdr e compare os resultados;
ANOVA no Rcmdr
- importe os dados apenas com as colunas de dados brutos;
- o menu
Statisticsestá separado em tipos de estatísticas e qual o parâmetro associado ao teste de hipótese estatístico; - o nosso teste é sobre médias, portanto no sub-menu
Mean; - nele há a opção
Multi-way ANOVA… - o resultado aparecerá na janela Output.
- 9) faça um gráfico que represente bem os dados;
- 10) interprete os resultados obtidos.
Exercício Anova
Delphinus nuttallianum
Vamos usar para esse exercício o exemplo do ótimo livro de estatística para ecólogos de Gotelli & Ellison (veja nossa lista de leituras recomendadas).
O experimento descrito analisou o efeito do degelo da primavera no crescimento e floração de uma planta alpina (Delphinus nuttallianum). Nesse experimento quatro parcelas foram mantidas sem nenhuma manipulação (unmanipulated), quatro foram aquecidas fazendo com que o degelo ocorresse antes do normal na primavera (treatment) e quatro foram manipuladas contendo toda a estrutura dos aquecedores, sem que estes fossem ligados (control). Os resultados do tempo de floração (dias) em cada parcela são apresentado abaixo:
| Unmanipulated | Control | Treatment |
|---|---|---|
| 10 | 9 | 12 |
| 12 | 11 | 13 |
| 12 | 11 | 15 |
| 13 | 12 | 16 |
- Organize esses dados em um planilha de forma que nas linhas estejam as observações e nas colunas as variáveis, no caso a reposta e preditora5). Para maiores informações sobre organização de dados em planilhas eletrônicas veja o artigo Data Organization in Spreedsheets (Broman & Woo, 2018)
- Construa a tabela de ANOVA e calcule o R² para esses dados em uma planilha eletrônica.
PARA ENTREGAR ANTES DO INÍCIO DA PRÓXIMA AULA:
Inclua os seguintes produtos no formulário abaixo:
1) Para os dados de solos e produtividade (Crawley, 2007):
- 1.1- a tabela de ANOVA completa gerada na planilha eletrônica;
- 1.2- a tabela de ANOVA resultante do teste no Rcmdr;
- 1.3- um gráfico para representar os resultados;
- 1.4- a interpretação dos resultados (máximo de 5 linhas).
2) Para os dados de Delphinus nuttallianum:
- 2.1- a tabela de ANOVA completa gerada em uma planilha eletrônica;
- 2.2- um gráfico para representar os resultados;
- 2.3- interpretação dos resultados desse experimento (máximo de 5 linhas)
- 2.4- a reposta para a seguinte questão: Por que o
unmanipulatednão é o controle para o tratamento que manipulou o degelo?(máximo 5 linhas)
Regressão Linear Simples
Conceitos importantes
Quando realizamos uma análise mais aprofundada sobre a relação entre duas variáveis numéricas contínuas podemos ajustar uma reta que represente o melhor ajuste entre os dados e que possa nos ajudar a prever valores da variável resposta (eixo Y) a partir de valores da variável preditora (eixo X). Se o ajuste é uma reta, esse tipo de análise é chamado de Análise de Regressão Linear. A explicação detalhada sobre como funciona essa análise foi apresentada na aula sobre Análise de Regressão Linear. Alguns aspectos importantes que precisam ser lembrados para entendermos esse tutorial:
- A reta ajustada (também denominada “linha de regressão”) passa obrigatoriamente pelo ponto que representa a média da variável Y e a média da variável X.
- Os pontos das observações estarão distribuídos em torno dessa reta.
- A relação (reta) entre a variável preditora (x) e a variável resposta (y) é descrita por uma equação simples (y=a+bx) com apenas dois parâmetros: a (intercepto) e b (inclinação da reta).
- O intercepto (a) representa o valor estimado da variável resposta (y) quando a variável preditora (x) é igual à zero.
- A inclinação (b) representa o efeito da variável preditora (x) sobre a variável resposta (y), ou seja, o quanto a variável y aumenta (ou diminui) à cada unidade da variável preditora (x).
- Quando usamos regressão linear para testar hipóteses científicas em ecologia, majoritariamente estamos interessados em saber o efeito da variável preditora (x) sobre a variável resposta (y), ou seja, se a inclinação da reta (b) é significativamente diferente de zero. Em geral, não temos hipóteses científicas acerca do intercepto do modelo.
- A distância vertical (projetada no eixo Y) de cada ponto até a reta é chamada de resíduo, desvio ou erro daquele ponto.
- A linha de regressão é aquela que minimiza os resíduos (na verdade, a soma dos quadrados dos resíduos)
O objetivo desse tutorial é fazer e interpretar os resultados de uma análise de regressão linear, assim como avaliar os pressupostos do modelo.
O que são os erros/resíduos e como calcular?
Os erros/resíduos indicam o quão longe os valores de Y observados estão dos valores de Y estimados pela linha de regressão ajustada. Eles estão representados em verde na figura abaixo:
Os erros/resíduos de cada observação são calculados projetando-se no eixo Y o valor de Y observado e o valor de Y estimado (ou predito) pela reta e calculando-se a diferença entre esses dois valores.
Premissas de uma Análise de Regressão Linear
- LINEARIDADE - Uma reta representa o melhor ajuste aos dados
- DISTRIBUIÇÃO NORMAL DOS ERROS/RESÍDUOS - Para cada valor de X, os erros devem seguir uma distribuição normal. Se fossem feitas muitas réplicas para cada um dos valores de X, a distribuição dos erros referentes aos vários valores obtidos para Y nas muitas réplicas seguiria uma distribuição normal (Veja as curvas em vermelho na figura de homoscedasticidade abaixo). Porém, em geral, não são feitas réplicas e é necessário assumir que os resíduos seguem essa distribuição.
- VARIÂNCIA DOS ERROS/RESÍDUOS CONSTANTE - Para qualquer valor de X, a variância dos erros é a mesma. Se fossem feitas muitas réplicas para cada um dos valores de X, a distribuição dos erros referentes aos vários valores obtidos para Y nas muitas réplicas apresentaria uma mesma variância para qualquer valor de X. Porém, em geral, não são feitas réplicas e é necessário assumir que essas variâncias são iguais.
Quando essa premissa é cumprida, temos o que chamamos de homoscedasticidade:

Quando ela não é cumprida, observamos uma heteroscedasticidade. Note na figura abaixo que para valores pequenos de X a variância é menor (distribuição estreita) e que para valores maiores de X temos uma variância grande (distribuição larga):

Checando as premissas
Ok, agora que você entendeu como funciona a regressão linear, vamos para um exemplo prático.
Baixe o arquivo de dados para o seu diretório:
- produtividade_chuva.txt|chuva.txt}}
Descrição dos conjuntos de dados:
Atenção: esses conjuntos de dados não são reais, são simulações produzidas com o objetivo pedagógico.
Pesquisadores interessados em entender o efeito da precipitação sobre a produtividade primária liquída em ecossistemas terrestres, selecionaram 30 áreas naturais distribuídas por todo o globo em diferentes ecossistemas. Dada a importância da água para a fotossíntese, a hipótese dos pesquisadores era que quanto maior a precipitação, maior seria a produtividade primária líquida do ecossistemas. Em cada área, os pequisadores coletaram duas informações: a precipitação anual média (mm) e a produtividade primária líquida (Mg/ha/ano).
Hipóteses estatísticas
Considerando que os pesquisadores estão interessados no efeito da precipitação sobre a produtividade, podemos assumir que a precipitação é a variável preditora (x) e a produtividade é a variável resposta (y). Como ambas as variáveis são contínuas, podemos aplicar uma regressão linear simples para testar a hipótese científica. Neste caso, o efeito de precipitação sobre a produtividade será descrito pela inclinação da reta (b). Sendo assim, as hipóteses estatísticas serão:
- H0: B=0
- H1: B≠0
Como saber se a variância dos erros/resíduos é constante?
Considerando que a reta obtida pelo modelo linear separa os pontos observados de Y de modo que eles fiquem distribuídos da melhor forma possível acima e abaixo da reta, teremos tanto valores positivos quanto valores negativos de resíduos para os diferentes valores de X.
Para um dado valor de X, teremos um valor de Y_estimado (que aparece na planilha de dados como “fitted.RegModel.*”). Relembrando, os Y_estimados são os valores projetados em Y quando o valor de X cruza a reta de regressão.
Se esperamos que a variância dos resíduos seja constante ao longo dos valores de X, deveríamos também esperar que o espalhamento dos valores dos resíduos (positivos ou negativos) sejam similares para os diferentes valores de Y_estimado.
Então, podemos fazer um gráfico em que relacionamos os valores de Y_estimado (“fitted.RegModel.*”) e os valores dos Resíduos (“residuals.RegModel.*”) para cada Y_estimado. Com esse gráfico podemos avaliar se a distribuição dos resíduos é similar ou se há um maior ou um menor espalhamento dos valores de resíduos para alguns valores de Y_estimado.
residuo2
Como você interpreta esse gráfico? Você nota algum padrão na distribuição dos erros/resíduos?
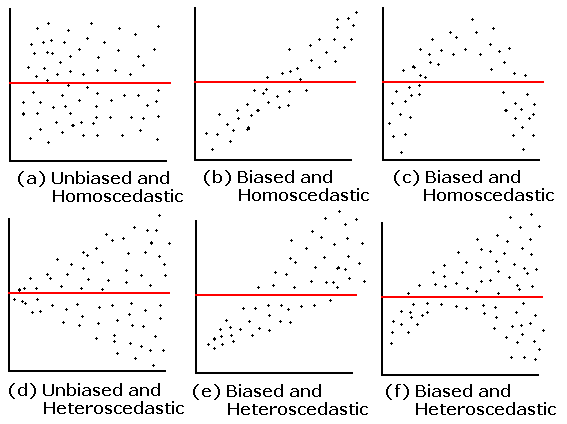
Esse mesmo gráfico que é utilizado para avaliar se a variância é constante (homoscedasticidade), também pode ser utilizado para checar se existe alguma assimetria, algum viés (positivo ou negativo) ou alguma tendência de que a relação seja melhor definida por uma curva do que por uma reta.
A figura abaixo mostra vários exemplos desse gráfico entre Resíduos e Y_estimado, com ou sem homoscedasticidade e com ou sem vieses (Biased ou Unbiased):
Ao interpretar esses gráficos, lembre-se sempre que aqui não estão sendo representados os seus dados brutos, e sim os resíduos e os valores de Y_estimado!
Como saber se alguma observação está influenciando demais os parâmetros da regressão?
Além de testar as premissas, também é importante fazer um diagnóstico para verificar se existem outliers e se eles influenciam muito o resultado da análise de regressão.
Para medir a influência que uma dada observação tem sobre a inclinação da reta estimada pelo modelo de regressão linear, usamos uma medida denominada “Distância de Cook” que é calculada para cada observação e avalia a relação entre o erro/resíduo (e) e a leverage (hii) da observação. A leverage (que pode ser traduzida como “alavancagem”) indica o quanto um dado valor de X é extremo considerando a amplitude dos demais valores de X. Repare, pela equação abaixo, que quanto maior for o erro/resíduo (e) e a leverage (hii) de uma dada observação, maior será a distância de Cook referente a ela, ou seja, sua influência sobre a estimativa dos parâmetros da distribuição. Porém, se a leverage for alta para uma dada observação, mas o erro/resíduo for pequeno, essa observação não terá um valor alto de Distância de Cook, ou seja, não terá tão grande influência sobre a inclinação da reta.
Valores altos de Distância de Cook para uma dada observação indicam que se ela fosse retirada das análises, a inclinação da reta de regressão poderia mudar muito. Veja o exemplo abaixo, do livro de Quinn & Keough (2008), mostrando o efeito de três diferentes observações sobre a inclinação da reta. Os números das retas (1, 2 e 3) indicam como seria a reta se aquela determinada observação (1, 2 ou 3, respectivamente) fosse mantida no conjunto de dados. A reta 1 indica como ficaria a reta sem as observações 2 e 3.
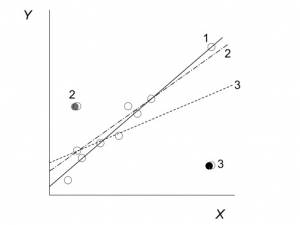
Se você não entendeu essa figura, peça ajuda!
Então, podemos fazer um gráfico em que plotamos o valor dos Resíduos em relação aos valores de leverage e nesse gráfico os pontos que possuírem as maiores leverage e os maiores erros/resíduos (positivos ou negativos) serão as observações com maiores Distâncias de Cook e consequentemente, com maiores influências sobre os parâmetros da reta.
Devido ao tempo escasso, não vamos construir esse gráfico passo-a-passo. Vamos usar uma opção mágica que vai mostrar 4 gráficos de diagnóstico de uma só vez e incluirá esse gráfico para que você possa analisar.
Final
Entendendo essa figura:
- Os dois gráficos à esquerda relacionam os resíduos aos valores de Y_estimado. Dentre esses, o gráfico inferior utiliza os resíduos padronizados6) para diminuir eventuais problemas com assimetria (skewness) nos dados. Em geral, basta checar um deles e já será possível identificar problemas de heteroscedasticidade e de viés nos resíduos. O que vemos em nossos dados? De maneira geral, o dados tem variância homogênea e não apresentam viés. No entanto, há um único dado (número 15) que possui resíduo muito maior do que as demais observações (provavelmente um outlier).
- O gráfico superior à direita é o gráfico quantil-quantil. Ele nos ajuda a identificar se os resíduos 7) se ajustam bem a uma distribuição normal (checagem da normalidade dos resíduos). Se os pontos desse gráfico estiverem bem próximos da linha diagonal (observem principalmente as extremidades), isso indica que os valores dos resíduos estão bem ajustados a uma distribuição normal. Se nas extremidades os pontos estiverem distantes da linha, a distribuição dos resíduos é assimétrica, apresentando caudas mais longas ou mais curtas, a depender da posição em que ocorrem esses pontos distanciados .. Este gráfico também indica que os resíduos se ajustam bem a uma distribuição normal, com exceção do dado 15 que apresenta uma valor mauito alto e “puxa” a cauda da distribuição, deixando-a mais longa à direita.
- O gráfico inferior à direita é o gráfico que mostra a relação entre resíduos (padronizados) e a leverage das observações. É nesse gráfico que podemos também conferir a Distância de Cook. As linhas vermelhas tracejadas indicam os limites para valores de distância de Cook que são considerados altos (acima de 0,5). Pontos localizados fora dessa linha tracejada são observações com alta Distância de Cook e que devem, portanto, ser analisados cuidadosamente. Repare que os pontos com as maiores Distâncias de Cook têm números que ajudam você a identificar a qual observação o ponto se refere. Conforme já indicado nos gráficos anteriores, aqui fica claro que o dado 15 é um outlier pois apresenta resíduo muito elevado. No entanto, esse gráfico indica que seu papel não é muito preocupante já que ele não é um dado influente (está dentro do limite aceito para a distância de Cook), provavelmente porque possui baixa alavancagem. Sendo assim, não é necessário removê-lo e refazer a análise para avaliar seu papel nos resultados.
PARA ENTREGAR ANTES DO INÍCIO DA PRÓXIMA AULA
Vcs agora continuarão a pesquisa sobre o efeito das condições ambientais na produtividade primária de ecossistemas terrestres. Suponha que vcs estão interessados em entender o efeito da temperatura média anual (°C) sobre a produtividade primária líquida (MgC/ha/ano). Dado que a temperatura média anual dos ecossistemas terrestres está diretamente relacionada ao comprimento da estação de crescimento, a hipótese científica a ser testada é se a temperatura tem efeito positivo sobre a produtividade. Utilize o conjuntos de dados produtividade_temp.txt8), faça a regressão linear simples, interprete os resultados e avalie o atendimento das premissas do modelo. Preencha o seguinte formulário para registrar suas respostas.
Histograma
hist(lm.algas.peixes$residuals)
Boxplot
boxplot(lm.algas.peixes$residuals)
Gráfico Quantil-Quantil
qqnorm(lm.algas.peixes$residuals) qqline(lm.algas.peixes$residuals)
Como saber se a variância dos erros/resíduos é constante?
Considerando que a reta obtida pelo modelo linear separa os pontos observados de Y de modo que eles fiquem distribuídos da melhor forma possível acima e abaixo da reta, teremos tanto valores positivos quanto valores negativos de resíduos para os diferentes valores de X.
Para um dado valor de X, teremos um valor de Y_estimado (que aparece na planilha de dados como “fitted.RegModel.*”). Relembrando, os Y_estimados são os valores projetados em Y quando o valor de X cruza a reta de regressão.
Se esperamos que a variância dos resíduos seja constante ao longo dos valores de X, deveríamos também esperar que o espalhamento dos valores dos resíduos (positivos ou negativos) sejam similares para os diferentes valores de Y_estimado.
Então, podemos fazer um gráfico em que relacionamos os valores de Y_estimado (“fitted.RegModel.*”) e os valores dos Resíduos (“residuals.RegModel.*”) para cada Y_estimado. Com esse gráfico podemos avaliar se a distribuição dos resíduos é similar ou se há um maior ou um menor espalhamento dos valores de resíduos para alguns valores de Y_estimado.
res.a.p<-lm.algas.peixes$residuals
yest.a.p<-lm.algas.peixes$fitted.values
plot(res.a.p~yest.a.p, xlab="Y estimado", ylab="Resíduos")
Como você interpreta esse gráfico? Você nota algum padrão na distribuição dos erros/resíduos?
Esse mesmo gráfico que é utilizado para avaliar se a variância é constante (homoscedasticidade), também pode ser utilizado para checar se existe alguma assimetria, algum viés (positivo ou negativo) ou alguma tendência de que a relação seja melhor definida por uma curva do que por uma reta.
A figura abaixo mostra vários exemplos desse gráfico entre Resíduos e Y_estimado, com ou sem homoscedasticidade e com ou sem vieses (Biased ou Unbiased):
Ao interpretar esses gráficos, lembre-se sempre que aqui não estão sendo representados os seus dados brutos, e sim os resíduos e os valores de Y_estimado!
Como saber se alguma observação está influenciando demais os parâmetros da regressão?
Além de testar as premissas, também é importante fazer um diagnóstico para verificar se existem outliers e se eles influenciam muito o resultado da análise de regressão.
Para medir a influência que uma dada observação tem sobre a inclinação da reta estimada pelo modelo de regressão linear, usamos uma medida denominada “Distância de Cook” que é calculada para cada observação e avalia a relação entre o erro/resíduo (e) e a leverage (hii) da observação. A leverage (que pode ser traduzida como “alavancagem”) indica o quanto um dado valor de X é extremo considerando a amplitude dos demais valores de X. Repare, pela equação abaixo, que quanto maior for o erro/resíduo (e) e a leverage (hii) de uma dada observação, maior será a distância de Cook referente a ela, ou seja, sua influência sobre a estimativa dos parâmetros da distribuição. Porém, se a leverage for alta para uma dada observação, mas o erro/resíduo for pequeno, essa observação não terá um valor alto de Distância de Cook, ou seja, não terá tão grande influência sobre a inclinação da reta.
Valores altos de Distância de Cook para uma dada observação indicam que se ela fosse retirada das análises, a inclinação da reta de regressão poderia mudar muito. Veja o exemplo abaixo, do livro de Quinn & Keough (2008), mostrando o efeito de três diferentes observações sobre a inclinação da reta. Os números das retas (1, 2 e 3) indicam como seria a reta se aquela determinada observação (1, 2 ou 3, respectivamente) fosse mantida no conjunto de dados. A reta 1 indica como ficaria a reta sem as observações 2 e 3.

Se você não entendeu essa figura, peça ajuda!
Então, podemos fazer um gráfico em que plotamos o valor dos Resíduos em relação aos valores de leverage e nesse gráfico os pontos que possuírem as maiores leverage e os maiores erros/resíduos (positivos ou negativos) serão as observações com maiores Distâncias de Cook e consequentemente, com maiores influências sobre os parâmetros da reta.
Devido ao tempo escasso, não vamos construir esse gráfico passo-a-passo. Vamos usar uma opção mágica que vai mostrar 4 gráficos de diagnóstico de uma só vez e incluirá esse gráfico para que você possa analisar.
summary (lm.algas.peixes)
Agora, vamos definir que sejam construídos os 4 gráficos de diagnóstico para esse modelo e que eles sejam colocados em uma mesma página:
par(mfrow=c(2,2)) plot(lm.algas.peixes) par(mfrow=c(1,1))
Entendendo essa figura:
- Os dois gráficos à esquerda relacionam os resíduos aos valores de Y_estimado. Dentre esses, o gráfico inferior utiliza os resíduos padronizados9) para diminuir eventuais problemas com assimetria (skewness) nos dados. Em geral, basta checar um deles e já será possível identificar problemas de heteroscedasticidade e de viés nos resíduos. O que vemos em nossos dados? De maneira geral, o dados tem variância homogênea e não apresentam viés. No entanto, há um único dado (número 15) que possui resíduo muito maior do que as demais observações (provavelmente um outlier).
- O gráfico superior à direita é o gráfico quantil-quantil. Ele nos ajuda a identificar se os resíduos 10) se ajustam bem a uma distribuição normal (checagem da normalidade dos resíduos). Se os pontos desse gráfico estiverem bem próximos da linha diagonal (observem principalmente as extremidades), isso indica que os valores dos resíduos estão bem ajustados a uma distribuição normal. Se nas extremidades os pontos estiverem distantes da linha, a distribuição dos resíduos é assimétrica, apresentando caudas mais longas ou mais curtas, a depender da posição em que ocorrem esses pontos distanciados .. Este gráfico também indica que os resíduos se ajustam bem a uma distribuição normal, com exceção do dado 15 que apresenta uma valor mauito alto e “puxa” a cauda da distribuição, deixando-a mais longa à direita.
- O gráfico inferior à direita é o gráfico que mostra a relação entre resíduos (padronizados) e a leverage das observações. É nesse gráfico que podemos também conferir a Distância de Cook. As linhas vermelhas tracejadas indicam os limites para valores de distância de Cook que são considerados altos (acima de 0,5). Pontos localizados fora dessa linha tracejada são observações com alta Distância de Cook e que devem, portanto, ser analisados cuidadosamente. Repare que os pontos com as maiores Distâncias de Cook têm números que ajudam você a identificar a qual observação o ponto se refere. Conforme já indicado nos gráficos anteriores, aqui fica claro que o dado 15 é um outlier pois apresenta resíduo muito elevado. No entanto, esse gráfico indica que seu papel não é muito preocupante já que ele não é um dado influente (está dentro do limite aceito para a distância de Cook), provavelmente porque possui baixa alavancagem. Sendo assim, não é necessário removê-lo e refazer a análise para avaliar seu papel nos resultados.
PARA ENTREGAR ANTES DO INÍCIO DA PRÓXIMA AULA
Vcs agora continuarão a pesquisa sobre o efeito das condições ambientais na produtividade primária de ecossistemas terrestres. Suponha que vcs estão interessados em entender o efeito da temperatura média anual (°C) sobre a produtividade primária líquida (MgC/ha/ano). Dado que a temperatura média anual dos ecossistemas terrestres está diretamente relacionada ao comprimento da estação de crescimento, a hipótese científica a ser testada é se a temperatura tem efeito positivo sobre a produtividade. Utilize o conjuntos de dados produtividade_temp.txt11), faça a regressão linear simples, interprete os resultados e avalie o atendimento das premissas do modelo. Preencha o seguinte formulário para registrar suas respostas.
## copie uma linha por vez:
algas.peixes2 <- read.csv("algas_peixes2.csv", sep=";")
head(algas.peixes2)
summary(algas.peixes2)
scatterplot(BIOMASSA_PEIXES_HERB2~BIOMASSA_ALGAS2, data=algas.peixes2)
lm.algas.peixes2<-lm(BIOMASSA_PEIXES_HERB2~BIOMASSA_ALGAS2, data=algas.peixes2)
summary (lm.algas.peixes2)
## copie as três linhas juntas:
par(mfrow=c(2,2))
plot (lm.algas.peixes2)
par(mfrow=c(1,1))
## copie uma linha por vez:
insetos.peixes <- read.csv("insetos_peixes.csv", sep=";")
head(insetos.peixes)
summary(insetos.peixes)
scatterplot(BIOMASSA_PEIXES_INS~BIOMASSA_INSETOS, data=insetos.peixes)
lm.insetos.peixes<-lm(BIOMASSA_PEIXES_INS~BIOMASSA_INSETOS, data=insetos.peixes)
summary(lm.insetos.peixes)
## copie as três linhas juntas:
par(mfrow=c(2,2))
plot (lm.insetos.peixes)
par(mfrow=c(1,1))
## copie uma linha por vez:
vol.inds <- read.csv("vol_inds.csv", sep=";")
head(vol.inds)
summary(vol.inds)
scatterplot(INDIVIDUOS_AUSTROL~VOLUME_LAGO, data=vol.inds)
lm.vol.inds<-lm(INDIVIDUOS_AUSTROL~VOLUME_LAGO, data=vol.inds)
summary(lm.vol.inds)
## copie as três linhas juntas:
par(mfrow=c(2,2))
plot (lm.vol.inds)
par(mfrow=c(1,1))