Ferramentas do usuário
Barra lateral
Tabela de conteúdos
Modelos Lineares Mistos
Esse roteiro é baseado em um material desenvolvido por alunos de nosso programa de PG 1)Melina Leite, Marília Gaiarsa e Lucas Medeiros e vem sendo modificado, ao longo dos anos, para adaptá-lo ao curso. Essa é a primeira versão utilizando o Rcmdr.
O objetivo desse roteiro é que você seja capaz de:
- 1. Entender o que são efeitos fixos e aleatórios.
- 2. Compreender a estrutura básica de um modelo linear misto.
- 3. Fazer uma análise de modelo misto no Rcmdr usando o pacote
lme4(Bates et al. 2014) - 4. Entender o output da função `lmer`.
- 5. Decidir quais efeitos aleatórios e fixos manter no seu modelo final.
- 6. Tirar conclusões a partir da análise de um modelo misto por meio de teste de hipótese ou seleção de modelos.
Modelos Mistos
Para explicar o que são modelos mistos e sua importância em ecologia, usaremos como exemplo um conjunto de dados presente no capítulo 5 de Zuur et al. (2009). Esses dados são referentes a um estudo sobre a riqueza de espécies da macrofauna em 9 praias na costa da Holanda. Em cada uma das praias os autores coletaram dados em cinco localidades diferentes. Para cada localidade existe informação sobre a altura da estação de amostragem em relação à altura média da maré (NAP, variável contínua) e também um índice de exposição da praia (Exposure, variável categórica).
Vamos supor que estamos interessados em verificar se a altura em relação à altura média da maré (variável NAP) influencia a riqueza de espécies nessas praias, deixando de lado a variável de exposição nesse momento. Podemos, por exemplo, construir um modelo linear simples da seguinte forma:
$$\text{riqueza} = \alpha + \beta * \text{NAP} + \varepsilon$$
sendo que $\varepsilon$ é um erro com distribuição normal (ou gaussiana) de média zero e um dado desvio padrão:
$$ \varepsilon = N(0, \sigma) $$
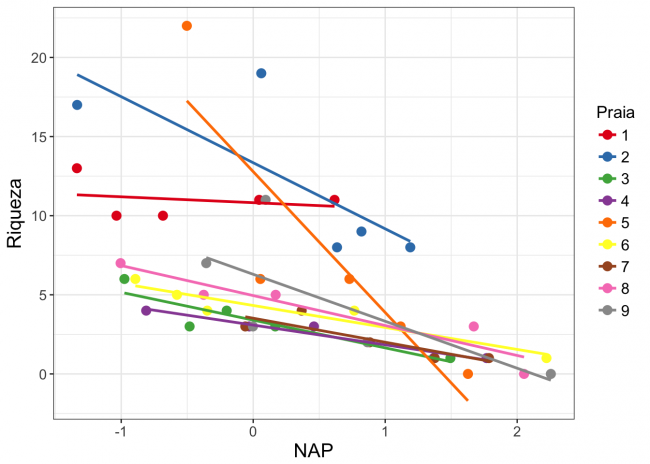
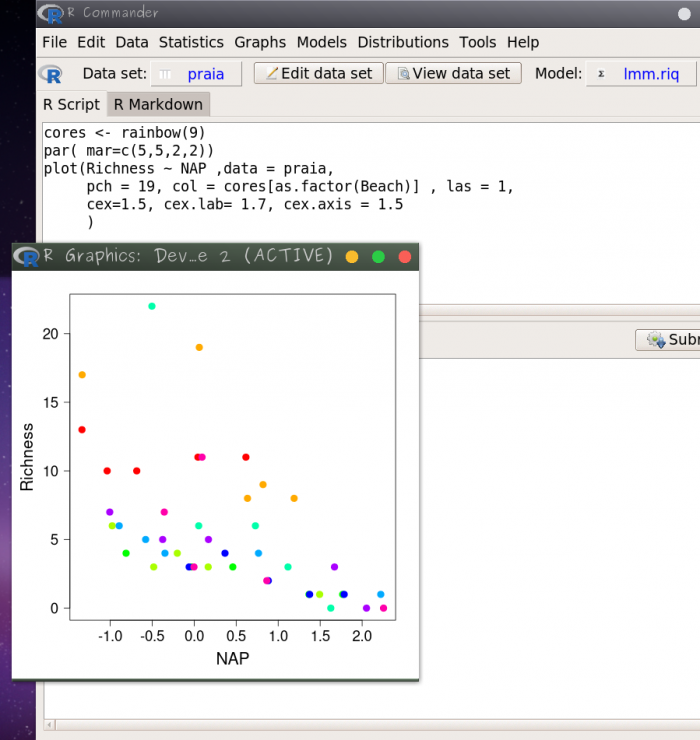
Aqui estamos modelando a riqueza de cada ponto de amostragem em função da variável NAP. O coeficiente $\alpha$ é o intercepto do modelo, $\beta$ é a inclinação (coeficiente angular ) de NAP e $\varepsilon$ é o resíduo do nosso modelo (erros), isto é, a variação da riqueza que não conseguimos explicar com nossa variável preditora NAP. No entanto, esse modelo tem um problema sério. Estamos violando uma premissa fundamental de modelos lineares, a de que os dados são independentes uns dos outros (Winter, 2013). Os dados obtidos em uma mesma praia não são independentes entre si (dependência espacial). Podemos imaginar diversas características de cada praia, como o tipo de grão de areia ou a força das ondas, que podem influenciar a riqueza de espécies e que não estão contempladas nesse modelo, podendo fazer com que os valores de riqueza obtido pelas unidades amostrais de uma mesma praia sejam mais parecidos entre si e que podem, ou não, ter relação com as variáveis preditoras que estamos analisando. Veja o gráfico abaixo, em que cada cor representa uma praia, para avaliar visualmente se a relação entre riqueza e NAP pode variar entre praias:
Nesta figura temos as retas do modelo entre Riqueza e NAP aplicado para cada praia (usando a função lm()). Entretanto, nossa pergunta inicial não diz respeito a cada praia separadamente, nós queremos modelar essa relação para todas as praias amostradas, sem especificar a relação para cada praia. Por outro lado, se considerarmos cada ponto de amostragem como se fossem independente entre si, estaremos incorrendo em um erro denominado de pseudo-replicação ou falta de independência das observações2). Ou seja, queremos considerar essa dependência sem falar especificamente de nenhuma praia e, ao mesmo tempo, avaliar a variação associada às praias. Além disso, queremos que as praias amostradas representem uma amostra aleatória de todas as praias que gostaríamos de contemplar no nosso estudo.
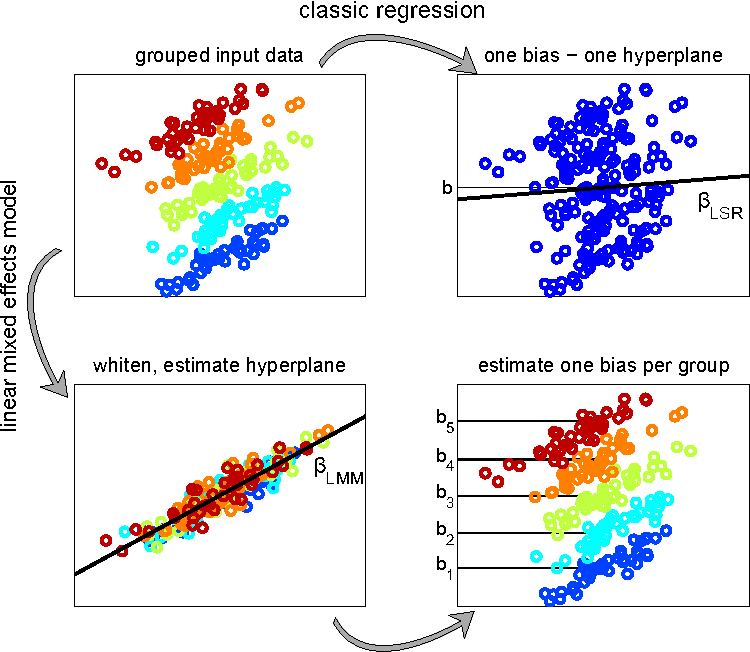
Efeito Aleatório
Para isso, precisamos de um modelo linear que incorpore o fato de que nossos dados estão agrupados em praias. Chamamos de efeito aleatório uma variável que agrupa nossos dados e que, geralmente, seu efeito sobre a variável resposta não nos interessa diretamente 3). Nesse exemplo, os autores amostraram nestas 9 praias, mas poderiam ter feito o mesmo estudo escolhendo outras praias. O efeito aleatório praia organiza parte da variação nos nossos dados que não conseguimos explicar, presente no erro $\varepsilon$ do modelo linear simples descrito no início desse roteiro (Winter, 2013). Por outro lado, as variáveis preditoras que estamos acostumados a encontrar em modelos lineares são chamadas de efeitos fixos. No exemplo das praias, a variável NAP é um efeito fixo e estamos diretamente interessados em seu efeito sobre a variável resposta. O nome misto refere-se ao fato de existirem tanto efeitos fixos, quanto aleatórios no modelo. Esses modelos são chamados também de “hierárquicos” ou “multinível”.
Em ecologia é comum encontrarmos delineamentos amostrais ou experimentais que geram dados com algum tipo de agrupamento (delineamento hierárquico ou aninhado). Por exemplo, quando uma amostragem é feita por parcelas ou quando um experimento é separado em diferentes blocos. Nesses casos, não podemos tratar nossos dados como independentes e a abordagem estatística mais adequada é a de modelos mistos.
Na seção a seguir veremos como explorar modelos mistos no R, usando a interface do Rmcdr. Inicialmente, a forma mais simples de incorporar o efeito aleatório em nosso exemplo é definir que cada praia apresenta um intercepto diferente no modelo. Ou seja, queremos ajustar uma reta para nossa relação entre riqueza e NAP, mas permitir que cada praia tenha seu próprio intercepto, que é relacionado ao intercepto da reta principal (efeito fixo).
Nosso modelo será:
$$\text{riqueza}_{i} = \alpha + \epsilon_{\text{praia}_i} + \beta * NAP + \epsilon_{\text{res}}$$
sendo:
$$\epsilon_{\text{praia}} ~ N(0, \sigma_{\text{praia}})$$
$$ \epsilon_{\text{res}} ~ N(0, \sigma_{\text{res}})$$
A riqueza da praia i é explicada pelo efeito fixo $\beta*NAP$ e pelo efeito aleatório $\epsilon_{\text{praia}_i}$, que se soma ao valor do intercepto fixo do modelo, $\alpha$, para formar o intercepto da praia i. Veja que o índice i está atrelado a $\epsilon_{\text{praia}_i}$ e, portanto, o modelo permite que cada uma das praias apresente um intercepto diferente com mesmas inclinações $\beta$. O $\beta$ faz parte do efeito fixo, e não possui nenhum índice i atrelado a ele, portanto é o mesmo para todas as praias. Finalmente, $\epsilon_{\text{res}}$ representa o resíduo associado aos desvios dos valores observados em relação aos preditos pela reta da praia relativa à observação.
Ambos, $\epsilon_{\text{praia}}$ e $\epsilon_{\text{res}}$, são variáveis aleatórias com média zero e desvios padrão $\sigma_{\text{praia}}$ e $\sigma_{\text{res}}$, respectivamente.
Mãos à obra! Modelos mistos no Rcmdr
Existem vários pacotes disponíveis no R para realizar análises de modelos mistos. Neste roteiro usaremos o lme4 (Bates et al. 2014), que possui funções para analisar modelos lineares mistos, modelos lineares mistos generalizados e modelos mistos não lineares. A seguir, vamos analisar os dados provenientes do capítulo 5 de Zuur et al. (2009) que exploramos na seção anterior.
Desde o ano de 2020, os modelos mistos passaram a ser contemplados no Rcmdr. Por isso, podemos utilizar essa interface para fazer os modelos nesse curso. Nos anos anteriores tivemos que utilizar os comandos diretamente no R. (Veja como o destino está ao seu lado!) Porém, lembre-se que vamos usar esse um pacote lme4 do Douglas Bates associado a essas análises. Então, ele precisa ser carregado quando o Rcmdr é aberto. Para isso, vá em Tools → load packages e escolha lme44).
Os dados estão disponíveis no site do livro do Zuur et al. (2009). A variável Exposure tem 3 níveis, sendo “8”, “10” e “11”, mas como o valor “8” foi observado apenas em uma praia, foi necessário reclassificar esta praia para o segundo valor mais baixo (Zuur et al. 2009).
Lendo os dados praias no R
- baixe o arquivo vamos à praia
- no Rcmdr, importe o arquivo, que está formatado com campos separados por tabulação e decimal como ponto
- atribua o nome de
praiaao conjunto de dados - visualize o conjunto de dados para se familiarizar com as variáveis
Ajustando os dados
A variável fExp está definida com os valores 10 e 11 para designar dois níveis de exposição das praias, mas como queremos que o Rcmdr interprete esses valores como categorias, precisamos transformar os valores 10 e 11 de fExp em fatores low e high, nesta ordem, e criando uma nova variável fExposure:
- abra o menu Data> Manage variables in active data set> Convert numeric variables to factors…
- coloque o nome da nova variável como
fExposureno campo<same as variable> - deixe selecionada a opção
Supply level names - no quadro que irá se abrir digite low para o valor 10 e high para o valor 11
- após criar verifique se a variável foi corretamente criada clicando no botão View data set
Estrutura do Modelo
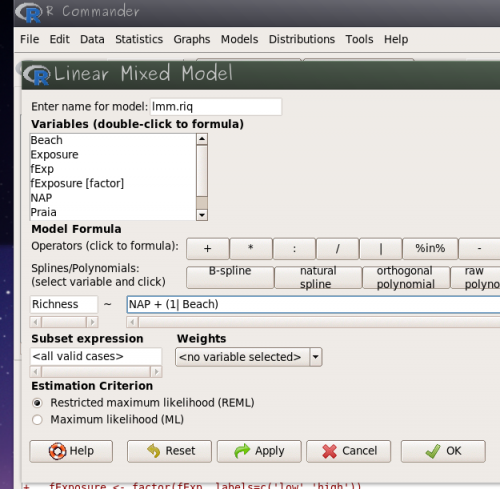
Agora, vamos construir nosso modelo. Relembrando, estamos interessados em ver se existe um efeito de NAP na riqueza de espécies. Nossos dados contam com nove diferentes praias e cinco diferentes pontos de amostragem para cada uma delas. Dessa forma, temos como efeito fixo a variável NAP, e como efeito aleatório a variável praia (Beach), que significa que cada praia terá um intercepto diferente. Assim, nosso modelo é:
Richness ~ NAP + (1 | Beach)
No Rcmdr siga os passos:
- abra o menu Statistics>Fit models>Linear mixed models…
- nomeie o modelo como
lmm.riqe coloque a fórmula acima nos campos adequados
Em seguida, vamos dar uma olhada no output do modelo que aparece na janela inferior do Rcmdr.
Interpretando o modelo
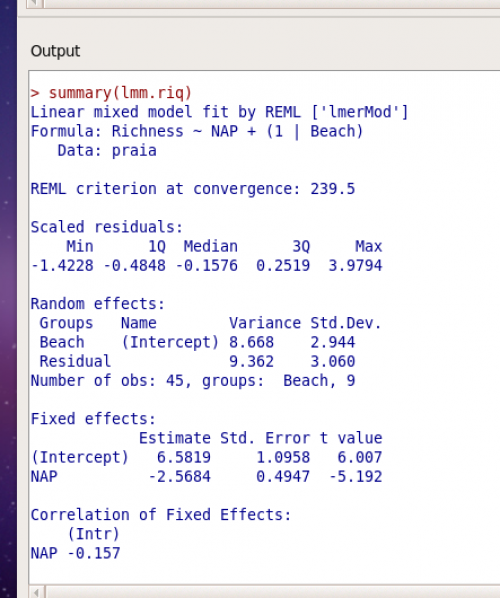
Na figura abaixo temos o resultado completo desse modelo lmm.riq:
Vamos começar olhando a parte dos efeitos aleatórios (Random effects). O desvio padrão (Std.Dev.) é uma medida do quanto a variabilidade da nossa variável dependente Richness é devida aos dois efeitos aleatórios que estamos analisando (os interceptos das praias e os resíduos). Podemos ver em Beach o desvio padrão associado às diferenças de intercepto entre praias ( $\sigma_{\text{praia}}$ do modelo associado ao $\epsilon_{\text{praia}}$). A última linha apresenta o resíduo, que indica o quanto da variabilidade não é prevista pela variável aleatória praia nem pela variável fixa NAP, que nada mais é que o $\sigma_{\text{res}}$ associado ao $\epsilon_{\text{res}}$ explicado anteriormente.
Os efeitos fixos indicam os coeficientes estimados para cada um dos fatores que estamos considerando como fixos. No caso, temos o intercepto (médio de todas as praias), que é a riqueza quando o NAP é zero (6.5819), e o coeficiente angular de NAP (-2.5684), o que indica que há uma diminuição de riqueza da ordem de mais de 2.5 espécies a cada unidade de NAP acrescida.
Note que nesse modelo a inclinação é a mesma para todas as praias e o intercepto médio dos coeficientes da estrutura aleatória do modelo é o intercepto da estrutura fixa.
Predito pelo Modelo
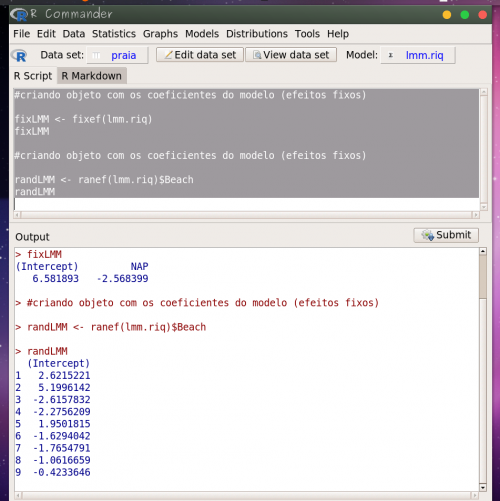
Para fazer o gráfico de nosso modelo ajustado, com a reta média predita pela estrutura fixa do modelo e as retas da estrutura aleatória, preditas para cada praia, vamos criar um objeto no Rcmdr com todos os coeficientes necessários. Copie as linhas de código a seguir no painel superior do Rcmdr (o painel chamado Rscript), em seguida, selecione todas as linhas copiadas e clique no botão Submit 5).
#criando objeto com os coeficientes do modelo (efeitos fixos) fixLMM <- fixef(lmm.riq) fixLMM #criando objeto com os coeficientes do modelo (efeitos fixos) randLMM <- ranef(lmm.riq)$Beach randLMM
O predito pela estrutura das variáveis fixas do modelo estimam o modelo médio, que seria a esperança do modelo para uma praia média, ponderada pelas observações de cada uma das praias em nosso dados. Além disso, a estrutura aleatória do modelo prediz um intercepto para cada praia das nossas observações e nos dá uma ideia da variação existente entre praias.
Note que nesse modelo temos apenas um coeficiente de inclinação, pois não modelamos a inclinação para a variável aleatória, apenas o intercepto. Mais adiante faremos um modelo incluindo inclinação para a variável aleatória e poderemos comparar os resultados.
Gráfico do Modelo
Vamos primeiro construir um gráfico base, com os valores observados. Para tornar o gráfico mais informativo vamos colocar cores para identificar de que praia a observação foi coletada. No código abaixo temos as seguintes sequência de eventos:
cores…: cria um objeto com nove cores tiradas de uma paleta das cores do arco-írispar(…): modifica parâmetros da janela gráfica, no caso abaixo modificamos as margens do gráficoplot(…): cria o gráfico- 1° linha: indica quais variáveis devem estar no eixo Y e X, e em qual o objeto estão os dados
- 2° e 3° linhas: modificações de parâmetros do gráfico, como símbolos, letras, etc
Então, copie os códigos abaixo para a janela superior do Rcmdr (RScript), selecione essas linhas e aperte o botão Submit:
cores <- rainbow(9)
par( mar=c(5,5,2,2))
plot(Richness ~ NAP ,data = praia,
pch = 19, col = cores[as.factor(Beach)] , las = 1,
cex=1.5, cex.lab= 1.7, cex.axis = 1.5
)
Incluindo o modelo no gráfico
Vamos usar os valores dos coeficientes (aqueles que calculamos anteriormente com as funções fixef e ranef) para representar as esperanças do modelo e criar as retas para cada praia e a reta média.
Primeiro o gráfico com as retas de cada praia:
Copie os códigos abaixo para a janela superior do Rcmdr (RScript), selecione essas linhas e aperte o botão Submit:
abline(randLMM[1,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[1]) abline(randLMM[2,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[2]) abline(randLMM[3,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[3]) abline(randLMM[4,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[4]) abline(randLMM[5,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[5]) abline(randLMM[6,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[6]) abline(randLMM[7,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[7]) abline(randLMM[8,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[8]) abline(randLMM[9,'(Intercept)']+ fixLMM[1], fixLMM[2], col=cores[9])
Para finalizar o gráfico, vamos colocar o predito pelo modelo médio e uma legenda:
Repita o mesmo procedimento de copiar, selecionar e “submeter”:
abline(fixLMM[1], fixLMM[2], col = "black", lwd=3, lty= 2)
legend("topright", c( "praia média",paste("praia", 1:9)) , lty=c(2, rep(1,9)), col= c(1,cores), bty="n", cex=1.2)
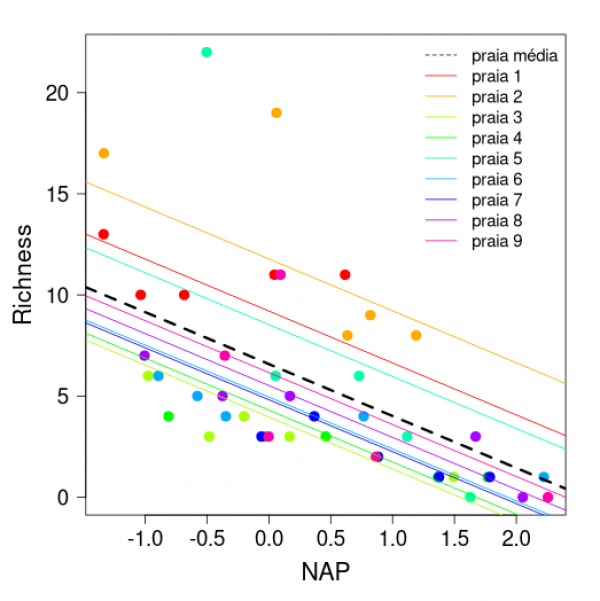
Ao final o nosso gráfico deve ser parecido com o que está a seguir:
Podemos ver nessa figura a predição do nosso modelo em relação aos parâmetros fixos (reta tracejada em preto) e as predições para cada praia separadamente. Como o efeito aleatório do nosso modelo estava apenas variando os valores de intercepto das praias, as retas para cada praia são paralelas.
Existem, entretanto, diversas possibilidades de modelos com esses mesmos dados. Por exemplo, podemos adicionar fExposure, que define o nível de exposição de cada praia, como uma variável preditora categórica e/ou podemos inserir o efeito aleatório da variável praia na inclinação da variável NAP (efeito fixo). Essa inclusão da interação entre a variável aleatória (praia) e a preditora fixa contínua (NAP) faz com que cada praia possa também ter uma relação específica com NAP (ou seja, inclinações de reta diferentes para cada praia), mas sempre ponderadas pela relação média.
Uma questão central em planejamento e análise de dados é partir de um conjunto de variáveis e termos para decidir como compor um conjunto de modelos plausíveis. A partir deles precisamos tomar uma decisão sobre qual é o melhor para ser interpretado. Aqui vamos continuar a usar o mesmo procedimento que usamos ao longo de todo o curso, ou seja, fazer o teste de hipótese para comparar modelos dois a dois a partir do modelo cheio para chegar ao mínimo adequado. A maior complexidade aqui é que os modelos mistos tem uma estrutura aleatória e uma fixa que devem ser investigadas.
Escolha dos efeitos aleatórios
Existem modelos e, portanto, perguntas e delineamentos amostrais que requerem apenas um efeito aleatório para indicar o agrupamento dos dados. Entretanto, como colocado no final da seção anterior, há também modelos que podem incluir mais de um efeito aleatório. Esse é o caso da interação entre praia e NAP mencionada acima. Se olharmos para o primeiro gráfico que fizemos para esse conjunto de dados 6), veremos que cada praia parece ter seu próprio intercepto e inclinação, o que torna plausível pensarmos que o modelo com a interação se ajuste melhor a esses dados.
Para modelos que podem ter mais de um efeito aleatório, Zuur et al. (2009), sugere um protocolo para a escolha da melhor estrutura de efeitos aleatórios. Vamos aos passos:
1. A primeira coisa a ser feita é ajustar um modelo com todos os efeitos fixos que estão sendo testados (modelo “completo”). No nosso exemplo tínhamos apenas NAP, mas vamos incluir fExposure na estrutura fixa e vamos incluir a interação entre NAP e fExposure para exemplificar melhor. No nosso modelo “completo” a estrutura fixa é:
Richness ~ fExposure + NAP + fExposure:NAP
2. Depois que criamos a estrutura fixa do nosso modelo “completo”, adicionamos os efeitos aleatórios plausíveis. No nosso exemplo, escolhemos duas possibilidades de efeito aleatório: i) apenas variações no intercepto entre praias ((1|Beach)); ii) contendo a interação entre praia e NAP, resultando também na variação de inclinação entre praias ((NAP|Beach)).
Poderíamos também ajustar um modelo sem efeito aleatório (usando a função lm) e ver se a inclusão do efeito aleatório resulta em um melhor ajuste do modelo, entretanto, a dependência entre as amostras de uma mesma praia são parte do desenho experimental dos dados e acreditamos que essa característica deve ser mantida no modelo. Além disso, é importante lembrar que a retirada de todos os efeitos aleatórios superestima os graus de liberdade do modelo.
Então, vamos lá! Primeiro começamos construindo o modelo lmmfull com todos os efeitos aleatórios, pelo menu do Rcmdr Statistic>Linear mixed models… e inserindo os termos do modelo como indicado abaixo:
Richness ~ fExposure + NAP + fExposure:NAP + (NAP|Beach)
Em seguida faça o mesmo procedimento para criar o modelo lmmint com a variável aleatória somente no intercepto:
Richness ~ fExposure + NAP + fExposure:NAP + (1|Beach)
Atenção aqui:
Um ponto importante é que esses modelos serão ajustados utilizando uma forma diferente de estimar os valores. Ao invés da máxima verossimilhança (ML - Maximum Likelihood), vamos usar a máxima verossimilhança restrita (REML - Restricted Maximum Likelihood). Isso acontece porque a ML é enviesada para as estimativas de variância desses modelos e a REML corrige esse viés. Na prática, nós não precisamos fazer nada, pois a função lmer do pacote lme4, utilizada pelo Rcmdr para ajustar os modelos, sempre utiliza a REML (argumento REML = TRUE) como padrão.
3. Vamos utilizar o comando anova como temos feito para fazer a seleção dos modelos aninhados, buscando sempre a simplificação do modelo ao mínimo adequado, usando o menu do Rcmdr Models>Hipothesis tests>Compare two models… e selecionando os modelos lmmint e lmmfull.
Muita atenção aqui:
Um problema com a interface do Rcmdr é que precisamos incluir o argumento refit = FALSE na função anova para garantir que o procedimento irá manter a estimativa por REML e não irá reajustar os modelos por ML antes de comparar os modelos. O padrão da função anova quando aplicada à comparação de dois modelos mistos é reajustar para ML. Como a interface não oferece essa opção precisamos editar a linha de comando do painel superior.
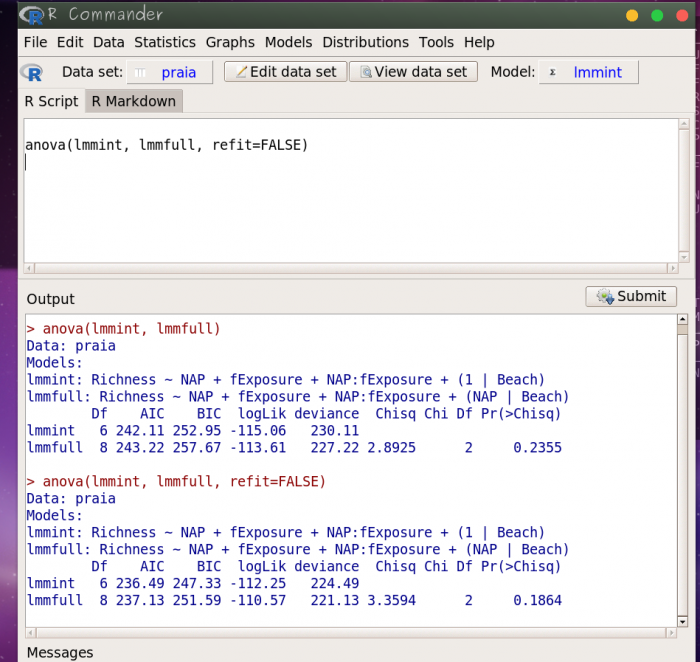
Quando você clicou ok na função anova de comparação de modelos, apareceu esse código na janela superior do Rcmdr:
anova(lmmint, lmmfull)
copie esse código novamente no painel superior, mas agora inclua a expressão refit = FALSE, como indicado abaixo
anova(lmmint, lmmfull, refit= FALSE)
Selecione essa linha e clique no botão Submit. O resultado deve ser como o apresentado abaixo:
Note que as estimativas do primeiro resultado (ou seja, da anova com reajuste para ML) e do segundo resultado (ou seja, sem reajuste para ML, usando refit = FALSE) são diferentes. É importante saber que no processo de simplificação da estrutura aleatória do modelo devemos ignorar os primeiros resultados, pois não queremos que seja feito um reajuste para ML, ou seja, queremos que a comparação das estruturas aleatórias seja feita usando as estimativas por REML.
O “p-valor” observado nessa comparação é alto, indicando que os modelos não apresentam diferenças marcantes, sendo assim, devemos reter o modelo mais simples e seguimos simplificando. Como só há mais um termo na estrutura randômica modelando o intercepto, podemos finalizar a seleção da estrutura randômica aqui.
Alguns autores (inclusive o próprio Zuur do capítulo que indicamos nesse tutorial como leitura) advogam que se nenhum termo da estrutura randômica for significativo, deve-se abandonar o modelo misto e partir para o modelo linear sem estrutura randômica. De fato, isso facilitaria muito a interpretação e a apresentação dos resultados do modelo. Porém, outros autores que seguiremos aqui, indicam que devemos manter a coerência do delineamento experimental/amostral e portanto devemos contemplar a dependência das observações de uma mesma praia, utilizando o modelo com a variável randômica praia no intercepto.
Bom, agora sabemos que a melhor estrutura de efeito aleatório é apenas a variação no intercepto entre as praias. Assim, podemos prosseguir com a verificação e simplificação dos efeitos fixos através de teste de hipóteses.
Note que quando observamos o primeiro gráfico desse tutorial, parecia que a inclusão de diferentes inclinações para cada praia no modelo seria importante. Porém, o resultado desse teste acima nos indica que, apesar de algumas praias terem inclinações aparentemente diferentes, não ganhamos muita informação adicional relevante ao incluir inclinações diferentes para todas as nove praias.
Inferência e diagnóstico do modelo
Nesta parte, depois de já escolhermos a estrutura aleatória do nosso modelo, podemos averiguar qual a real influência dos efeitos fixos sobre a riqueza de espécies, utilizando o procedimento que temos adotado nessa disciplina, ou seja o teste de hipóteses por meio da comparação de modelos pela tabela de ANOVA. Depois de selecionarmos o modelo mínimo adequado, que melhor se ajusta aos dados, vamos fazer o diagnóstico dos resíduos deste modelo para ver se ele atende às premissas de um modelo linear misto.
Teste de hipótese
O procedimento para verificar se termos do modelo são ou não significativos é a construção de modelos aninhados (ou seja, retirando um parâmetro do modelo com mais parâmetros) e a comparação por uma tabela de anova.
Muita atenção aqui:
No caso da comparação da estrutura fixa de modelos mistos com mesmo efeito aleatório precisamos reajustar nossos modelos para que as estimativas sejam feitas por ML (Máxima Verossimilhança). Mas, relembrando o que já foi dito anteriormente, a função anova, por padrão, já reajusta os modelos para ML quando compara dois modelos. Portanto, não precisamos nos preocupar em adicionar nenhum termo, mas é importante saber que esses procedimentos estão sendo feitos.
Então vamos começar usando nosso modelo cheio lmmint na comparação.
llmmint:
Richness ~ fExposure + NAP + fExposure:NAP + (1|Beach)
Vamos agora construir o modelo sem interação entre fExposure e NAP, denominando-o de lmm01.
Depois, comparamos esses modelos com o teste de hipótese por meio da função anova pelo menu do Rcmdr Models>Hipothesis tests>Compare two models…. E assim por diante, simplificando os modelos como já feito nos roteiros anteriores até chegar ao modelo mínimo adequado. Importante lembrar que a estrutura aleatória deverá ser sempre a mesma em todos os modelos que serão comparados!
Após definir o modelo mínimo adequado, analise o “summary” do modelo e avalie:
- na estrutura aleatória o valor do desvio padrão da variável aleatória é maior, menor ou equivalente ao desvio padrão dos resíduos?
- o que essa comparação representa?
- o que está representado no intercepto da estrutura fixa do modelo?
- qual a interpretação biológica dos coeficientes dos parâmetros da estrutura fixa do modelo?
Diagnósticos dos modelos
Depois de selecionado o modelo que melhor se ajusta aos nossos dados, precisamos avaliar o ajuste deste modelo a suas premissas. O Rcmdr faz alguns gráficos diagnósticos e de efeito do modelo que podem ser explorados no menu: Models>Graph>.
Para a construção de outros gráficos diagnósticos primeiro vamos carregar o pacote lattice selecionando-o pelo menu Tools>Load package(s)….
Depois, copie os códigos abaixo no painel superior (RScript) do Rcmdr, MAS, antes de apertar o botão “Submit” substitua o argumento NOME_DO_MODELO pelo nome do seu modelo mínimo adequado. Depois disso pode selecionar e submeter as linhas de comando.
Atenção: Faça um gráfico de cada vez e salve o pdf de cada gráfico antes de começar a fazer o próximo
## 1) gráfico quantil-quantil (normalidade) qqmath(NOME_DO_MODELO,id=0.05)
## 2) gráfico de valores ajustados x resíduos (homocedasticidade)
plot(NOME_DO_MODELO,type=c("p","smooth"))
## 3) gráfico de valores ajustados x resíduos padronizados (homocedasticidade)
plot(NOME_DO_MODELO,sqrt(abs(resid(.)))~fitted(.), type=c("p","smooth"))
OPS! Olhando o gráfico diagnóstico dos resíduos, parece que os dados não são tão homocedásticos como deveriam, pois vemos algo parecido com um funil se abrindo da esquerda para a direita, o que indica que o modelo viola esta premissa.
E agora??! Bem, quase sempre existe um caminho! O problema aqui é que assumimos que a riqueza de espécies, uma variável de contagem, poderia ser modelada como uma distribuição normal. Entretanto, dados de contagem geralmente são melhor modelados usando a distribuição de Poisson. Já aprendemos isso no tutorial Modelos Lineares Generalizados: contagem.
Então, para fazermos a modelagem correta dos nossos dados teremos que usar um modelo com distribuição de Poisson dentro do contexto de modelo misto. Nesse caso teríamos que usar uma classe de modelos que junta o GLM com o LMM que se chama Modelo Generalizado Misto.
Mas isso é tema para outro roteiro… Modelos Generalizados Mistos (GLMM)
Cultivando aveia
Nesse exemplo vamos usar os dados oats do pacote MASS.
- Carregue o pacote MASS através do menu Tools>Load packages…
- Entre no menu Data>Data in packages>Read data set on attached package…
- Selecione: na janela Package MASS e na Data set oats
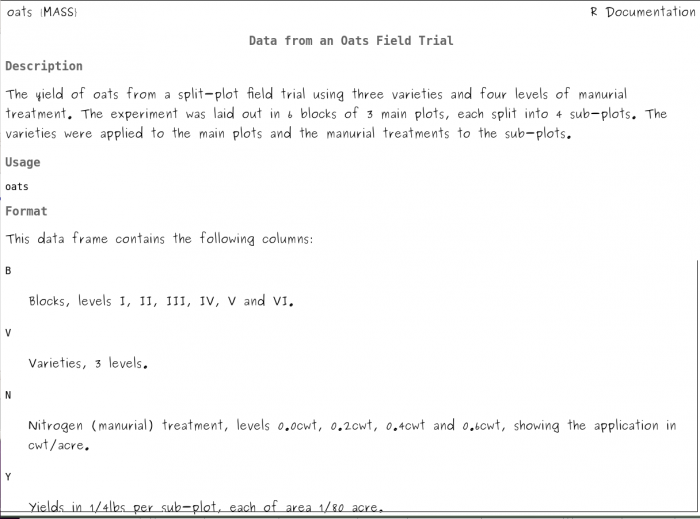
Para entender os dados utilize o o menu Help>Help on active data set (if available). Nessa documentação a descrição dos dados é:
Ajustando os dados de Aveia
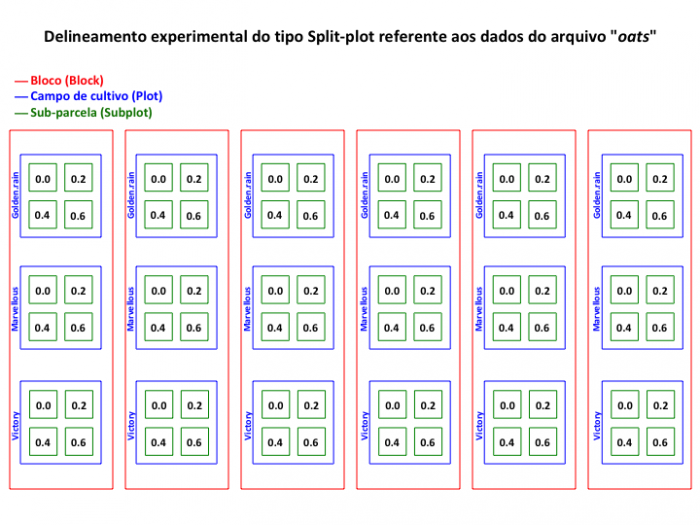
Como nesse caso os blocos são divididos em três campos de cultivo (plot) para cada variedade de aveia e cada campo é dividido em quatro partes (subplot) com os diferentes tratamentos com adubo nitrogenado, temos um aninhamento de bloco com plot e subplot. Como pode ser verificado na figura abaixo:
As variáveis plot e subplot não estão nesse conjunto de dados, mas podem ser resgatadas pelo fato de plot ser o campo onde foi cultivada cada variedade. Só precisaremos criar a variável plot, pois a subdivisão dos campos de cultivo nos diferentes níveis de adubação (que seriam os subplots) não precisa ser indicada no modelo, uma vez que essa divisão representa o menor nível hierárquico e a função lmer já reconhece esse nível automaticamente.
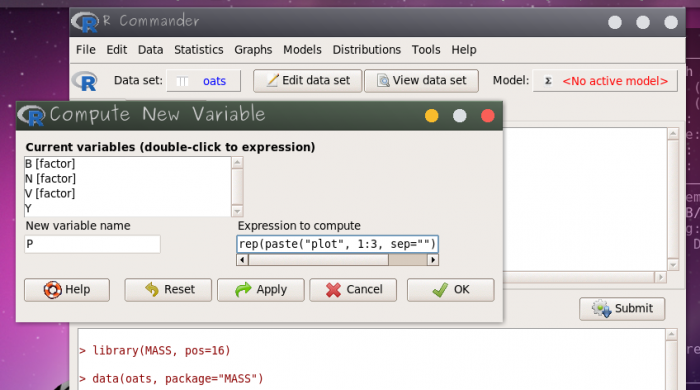
Para criar plot vamos usar o menu Data>Manage variable in active data set>Compute new variable e utilize o nome P (plot) no campo New variable name e no campo Expression to compute insira o código abaixo:
rep(paste("plot", 1:3, sep=""), each=4)
Esse código irá criar uma nova variável P com os níveis plot1, plot2 e plot3 para cada campo de cultivo de cada variedade de aveia, nos quais os níveis de adubação estão incluídos (4 níveis). Apesar da variável P estar no mesmo nível da variável V, fazemos isso para explicitar que P é diferente de V, sendo que uma representa os campos de cultivo e a outra representa as variedades de aveia cultivadas, respectivamente. Note também que os códigos de P se repetem entre os blocos, apesar de serem unidades distintas, ou seja, o campo de cultivo plot1 do bloco I é outro campo de cultivo do que o plot1 do bloco II. Por outro lado, as variedades se repetem entre os blocos, ou seja, a variedade Marvellous é Marvellous em qualquer bloco.
Atividade
- Construa um modelo cheio tendo como variável resposta o rendimento de cultivo (
Y) e como preditoras as variáveis fixas variedade (V) e adubação nitrogenada (N). Como estrutura aleatória utilize o fator aleatório plot (P) aninhado dentro de bloco (B)7). Note que como não temos variáveis fixas contínuas, apenas categóricas, não precisamos modelar a inclinação do modelo, apenas o intercepto. - Faça a simplificação da estrutura fixa das preditoras e chegue ao modelo mínimo adequado.
- Faça a interpretação biológica do resultado baseado nos coeficientes fixos e da variação associada aos fatores aleatórios.
Se você se interessou pelos modelos mistos e acha que eles se encaixam no seu problema, não deixe de conferir as referências que a gente listou abaixo para se aprofundar nesse universo!!
Exercício
PARA ENTREGAR ANTES DA PRÓXIMA AULA Acesse o formulário e responda às questões propostas referentes aos dois exemplos (Praias e Aveia) trabalhados nesse tutorial.
Referências e recomendações
Bates, et al. 2014.**Fitting linear mixed-effects models using lme4**. arXiv preprint arXiv:1406.5823. (publicação do pacote `lme4`)
Burnham, K. & Anderson, D. 2002.](http://gen.lib.rus.ec/book/index.php?md5=0572C2F65088CFA05EC3757297DBC173) Model selection and multimodel inference: a practical information-theoretic approach. 2nd edn. New York: Springer-Verlag. (Livro sobre a abordagem de seleção de modelos baseada em Teoria da Informação)
McGill, B. 2015. **Is it a fixed or random effect?** Blog Dynamic Ecology. (Uma boa discussão sobre o que são efeitos fixos e aleatórios)
Winter, B. 2013.[http://arxiv.org/pdf/1308.5499.pdf| Linear models and linear mixed effects models in R with linguistic applications. arXiv:1308.5499.]] (Nesse excelente roteiro, o autor explica modelos lineares e depois apresenta modelos mistos de uma forma bem didática)
Zuur, A., Ieno, E., Walker, N., Saveliev, A. & Smith, G. 2009. Mixed effects models and extensions in ecology with R. (Livro muito bom e completo sobre modelos mistos e aditivos)
R diretamente, você precisará instalar e carregar esse pacote# não são lidas pelo Rcmdr e podem ser usadas para comentar as linhas de código, isso ajuda a lembrar o que a linha de código faz(1|B/P)